ZNF226
Protein 226 cinkovog prsta je protein koji je kod ljudi kodiran genom ZNF226.[5]



Gen
urediProtein 226 cinkovog psta, također je znan i kao Kruppel-asocirani homeoboksni protein.[6] Kod ljudi, gen ZNF226 nalazi se na plus lancu hromosoma, pozicija 19q13, obuhvatajući 13.311 nukleotida od 44,165.070. do 44,178.381.[5]
Transkript
urediPostoji 20 različitih varijanti transkripta koji kodiraju ZNF226.[5] Svi oni imaju šest ili sedam identificiranih egzonskih regija unutar ZNF226.[7] Najduži identificirani transkript, varijanta transkripta ZNF226 x4, obuhvata 2.797 parova baza (bp).[7]
Protein
urediZna se da kod ljudi ZNF226 ima tri izoforme : ZNF226 izoforma X1, ZNF226 izoforma X2 i ZNF226 izoforma X3.[5] Proteinska izoforma X1 je najduža poznata varijanta, sa 803 aminokiseline.[5] Ovaj protein sadrži domen Krupekovog pridruženog boksa A (KRAB-A), koji funkcionira kao transkripcijski represor.[8] Međutim, tačna funkcija proteina ZNF226 još nije poznata. Unutar izoforme X1 postoji 18 domena C2H2 cinkovog prsta domena strukturnog motiva (zf-C2H2), za koje se zna da vežu bilo ione cinka (Zn2+) ili nukleinsku kiselinu (slika 7-8).[9][10] Unutar tih regija, cistein i histidin su primarne aminokiseline koje se vežu za Zn2+ ili nukleinsku kiselinu, iako su identificirane i druge aminokiseline za vezivanje (slika 7– 8). Pored domena KRAB-A i zf-C2H2, postoje i dvostruki domeni cinkovog prsta, koje sadrže i mjesta vezanja za ione ili nukleinske kiseline.[7]
ZNF226 sekvence ljudskih i ortoloških proteina imaju molekulsku težinu između 89 i 92 kDa.[11] Imali su izoelektrične tačke (pI) u rasponu od 8,60 do 9,00. U ljudi je regija dvostrukog domena zf-C2H2 i cinkovog prsta ZNF226 izoforme X1 59,3 kDa s teorijsim pI od 9,11.[11] Sa razmičućim cisteinom, ili C, postoji cistein sa svake tri aminokiseline. Najmanje jedna od aminokiselina između C su ili asparaginska ili glutaminska kiselina.[12] Uprkos obrascima asparaginske kiseline u regiji, i dalje se smatra da ima manju količinu ove aminokiseline od 1,9%. Postoji i ponavljanje unutar hemijskih obrazaca kod ljudi, što je karakteristično za ZNF226. Ova ponavljanja najčešća su unutar dvostrukih domena zf-C2H2 i proteina cinkovih prstiju, posebno sa veznim mjestima cisteina i histidina.[12] Predviđene sekundarne strukture ZNF226 pokazuju promjenjivi broj alfa-heliksa, beta-listova i slučajne zavojnice kroz protein. Korištenjem različitih programa, kao što su GOR4 i Chou i Fasmanov program, nađeno je da postoji ukupna sličnost u predviđanjima upredenih, usukanih i spiralnih regija u čitavom proteinu.[13][14][15]
- Aminokiselinska sekvenca
| 10 | 20 | 30 | 40 | 50 | ||||
|---|---|---|---|---|---|---|---|---|
| MNMFKEAVTF | KDVAVAFTEE | ELGLLGPAQR | KLYRDVMVEN | FRNLLSVGHP | ||||
| PFKQDVSPIE | RNEQLWIMTT | ATRRQGNLGE | KNQSKLITVQ | DRESEEELSC | ||||
| WQIWQQIAND | LTRCQDSMIN | NSQCHKQGDF | PYQVGTELSI | QISEDENYIV | ||||
| NKADGPNNTG | NPEFPILRTQ | DSWRKTFLTE | SQRLNRDQQI | SIKNKLCQCK | ||||
| KGVDPIGWIS | HHDGHRVHKS | EKSYRPNDYE | KDNMKILTFD | HNSMIHTGQK | ||||
| SYQCNECKKP | FSDLSSFDLH | QQLQSGEKSL | TCVERGKGFC | YSPVLPVHQK | ||||
| VHVGEKLKCD | ECGKEFSQGA | HLQTHQKVHV | IEKPYKCKQC | GKGFSRRSAL | ||||
| NVHCKVHTAE | KPYNCEECGR | AFSQASHLQD | HQRLHTGEKP | FKCDACGKSF | ||||
| SRNSHLQSHQ | RVHTGEKPYK | CEECGKGFIC | SSNLYIHQRV | HTGEKPYKCE | ||||
| ECGKGFSRPS | SLQAHQGVHT | GEKSYICTVC | GKGFTLSSNL | QAHQRVHTGE | ||||
| KPYKCNECGK | SFRRNSHYQV | HLVVHTGEKP | YKCEICGKGF | SQSSYLQIHQ | ||||
| KAHSIEKPFK | CEECGQGFNQ | SSRLQIHQLI | HTGEKPYKCE | ECGKGFSRRA | ||||
| DLKIHCRIHT | GEKPYNCEEC | GKVFRQASNL | LAHQRVHSGE | KPFKCEECGK | ||||
| SFGRSAHLQA | HQKVHTGDKP | YKCDECGKGF | KWSLNLDMHQ | RVHTGEKPYK | ||||
| CGECGKYFSQ | ASSLQLHQSV | HTGEKPYKCD | VCGKVFSRSS | QLQSHQRVHT | ||||
| GEKPYKCEIC | GKSFSWRSNL | TVHHRIHVGD | KSYKSNRGGK | NIRESTQEKK | ||||
| SIK |
- Simboli
Regulacija
urediGen
urediPromotor
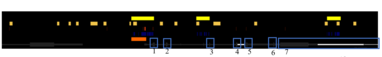
urediUpotrebom softvera Genomatix, GXP_7536741 (1142 bp) prepoznat je kao najbolji promotor ZNF226 (slika 1).[16] Unutar zadnjih 500 promotorskih bp, pretvarač signala i aktivator transkripcije (V $ STAT.01), faktor aktiviranja tRNK selenocisteina (V $ THAP11.01) i regulatori ćelijskog ciklusa: regije homologije ćelijskog ciklusa (V $ CHR. 01) su konzervirane među Homo sapiens, Macaca mulatta, Pan troglodytes, i Canis lupus familiaris.[16] Pored toga, proto-onkogen SPI-1, hematopoetski TF PU.1 (V $ SPI1.02) je također bio poznat po vezanju za promotorsku regiju unutar c-fes proto-onkogena, koji kodira enzim tirozin-kinaza.[17] Mjesto vezanja za TF također se nalazi u dvije regije unutar promotorske sekvence. Pretvarač signala i aktivator mjesta transkripcijskog vezanja, također su konzervirani u dvije regije, a poznato je da imaju veću specifičnost vezanja.[18] Selenocisteiski faktor za aktiviranje tRNK ima ulogu u regeneraciji embrionskih matičnih ćelija.[19] Regulatori ćelijskog ciklusa: vezno mjesto regije homologije ćelijskog ciklusa ima važnu ulogu u preživljavanju ćelija, gdje mutacije transkripcijskog faktora mogu dovesti do apoptoza.[20]
Tkivna distribucija
urediŠto se tiče ekspresije gena, ZNF226 je općenito eksprimiran u većini tkiva.[5] Podaci o mikromreži ilustriraju veću ekspresiju ZNF226 unutar jajnika.[21] To dodatno potkrepljuju podaci koji pokazuju smanjenje ekspresije ZNF226 u ćelije granuloze kod osoba sa sindromom policistastih jajnika.[22] Također je uočena jača ekspresija ZNF226 u štitnjači, u odnosu na druga tkiva.[5] Dokazi o smanjenoj ekspresiji ZNF226 uočeni su kod osoba sa papilskim karcinomom štitnjače.[23]
Unutar fetusa postoji određeni nivo ekspresije ZNF226 prisutan u svim tkivima tokom gestacijskog perioda od 10 do 20 sedmica.[5] Međutim, postoji i viši nivo ekspresije ZNF226 u srcu u 10 sedmica gestacije i smanjenog nivoa ekspresije u bubrezima u 20 sedmica gestacije.[5]
Ekspresija ZNF226 uočena je unutar epitelnih matičnih ćelija (EPC) u perifernoj krvi (PB) i krv iz pupkovine (CB). Ekspresija gena je niža u PB-EPC, u odnosu na CB-EPC-ove.[24] PB-EPC imaju jaču ekspresiju tumorskog supresora (TP53), u odnosu na CB-EPC.[24] CB-EPC imaju više angiogenih ekspresija ili rasta i cijepanje vaskulature.[24]
Transkript
urediKoristeći RNAfold, minimalne slobodne energije, stvorene su strukture na osnovu proširenih 5’i 3’ netranslatiranih regija (UTR) u ljudskim sekvencama. Nekonzervirane aminokiseline, miRNK, formacije matične petlje i RNK-vezujući proteini (RBP) prikazani su na dijagramu (slike 2–3).
Targetiranje miRNK
urediUnutar 5 ’UTR regije, i miR-4700-5p i miR-4667-5p su referencirane u eksperimentu koji je identificirao određene miRNK dosljedno eksprimirane u ERBB2+, genu za rak dojke.[25] Pored toga, na miR-8089 se navodi referenca u studiji koja pokazuje određene nove miRNK, pronađene u pacijenata sa sepsom.[26] Pokazalo se da miR-4271 ima efekte na koronarnu bolest srca vezujući se za 3 'UTR regiju gena APOC3.[27] Literatura o miR-7113-5p pokazuje da je ova miRNK mirtron.[28]
Unutar regije 3 'UTR, na miR-3143 se govori u studiji u kojoj su miRNK dosljedno eksprimirane u genu za ERBB2 + rak dojke.[25] miR-152-5p ima ulogu u inhibiciji gena za metilaciju DNK, koji su uključeni u metaboličke i upalne puteve.[29] miR-31-3p je prekomjerno eksprimiran u pločastoćelijskom karcinomu jednjaka (ESCC).[30] Jedan od rezultata miRNK, miR-150-5p, konzerviran je na više homologa unutar 3 ’UTR regije više od 3000 bp nizvodno.[31] miRNK miR-150-5p ima ulogu u kolorektumskom karcinomu (CRC), gdje je niža ekspresija miRNK povezana sa suzbijanjem metastaza u CRC-u.[31]
Proteini koji vežu RNK
urediŠto se tiče nekih pronađenih RBP-ova, PAPBC1 imao je pet mjesta vezanja, od kojih su dva istaknuta na 5 ’UTR. Poznato je da ovaj protein veže poli-a-repove za proteine koji su ušli u citoplazmu, sprečavajući njihov ponovni ulazak u jedro. Još jedan protein FUS pronađen je s vezivnim mjestom na predviđenoj petlji peteljke. Gen kodira protein koji olakšava transport proteina u citoplazmu.[32] Unutar 3 ’UTR-a, proteini RBMY1A1, RBMX i ACO1 su neki od najboljih RBP-ova. RBMY1A1 je protein za koji je poznato da sudjeluje u preradi i potreban je za razvoj sperme.[33] Protein RBMX je homolog proteina RBMY koji je uključen u proizvodnja sperme.[34] Također je poznato da promovira transkripciju gena za supresiju tumora TXNIP.[34] ACO1 je još jedan RBP za koji se zna da se veže sa iRNK za regulaciju nivoa gvožđa.[35] Vezivanjem za elementi koji reaguju na gvožđe, on može potisnuti translaciju feritina i inhibirati degradaciju prenosne mRNA receptora kada nivo gvožđa postane nizak.[35]
Protein
urediProvedene su analize za predviđanje posttranslacijskih modifikacija proteina. Na osnovu rezultata Expasyjevog miristoilatora kod Homo sapiens, južnog tuljana Mirounga leonina i krtičastog pacova Fukomys damarensis , može se zaključiti da ZNF226 nije miristiliran na N-kraju.[36] Brojna predviđena mjesta vezanja za posttranslacijske modifikacije također su identificirana među tri vrste. Područje fosforlacije na C-terminusu proteina također je identificirano kao podudaranje za mjesto vezanja fosforilaciju protein-kinaze C.[37][38][39][40] S-nitrozilacija je još jedna identificirana modifikacija na C354 (slika 2). Ova modifikacija pronađena je u SRG1, proteinu cinkovog prsta koji ima ulogu u sprečavanju sinteze dušičnog oksida (NO).[41] When NO is sustained, s-nitrosylation occurs within the protein, disrupting its transcriptional repression abilities.[41] Acetilacija je još jedna identificirana modifikacija. U slučaju promijelocitne leukemije, stanja koje rezultira obiljem krvotvornih ćelija u koštanoj srži, poznato je da u promijelocitnoj leukemiji, proteini cinkovog prsta aktiviraju histon-acetiltransferaze ili acetilaciju lizinskog C-kraja.[42] Poznato je da acetilacija u drugim proteinima cinkovog prsta, poput GATA1, poboljšava njihovu sposobnost interakcije s drugim proteinima.[43] Dimetilacija arginina je još jedna identificirana modifikacija unutar ZNF226. Pokazalo se da metilacija argininskog ćelijskog proteina koji veže nukleinsku kiselinu (CNBP), protein cinkovog prsta, ometa njegovu sposobnost vezanja nukleinskih kiselina.[44]
Predviđa se da se ZNF226 lokalizira unutar jedra, što se poklapa sa poznatim funkcijama kao faktora transkripcije.[45] Također se predviđa lokalizacija unutar mitohondrija.[45]
Homologija/evolucija
urediIako je malo dostupnih informacija o genu ZNF226, homolozi gena pronađeni su kod eukariotskih i bakterijskih vrsta. Strogi ortolozi pronađeni su samo kod sisara (slika 4). Gen ZNF226 je također usko povezan s paralogom ZNF234 kod ljudi i zfp111 genom kod miševa.[5][8] Među raznim vrstama kod kojih su identificirani ortolozi i homolozi ZNF226, očigledno su konzervirana vezna mjesta za C2H2 (slika 4-5).[12] U ljudskim paralozima ZNF226, postoji i konzervacija mjesta vezianja za C2H2, kao i mjesta vezanja nukleinske kiseline.[5]
Za protein ZNF226 vidljiv je sporo razvijanje. Razvija se na način sličan proteinu citohrom c, umjesto proteinu fibrinogenogenskog alfa lanca (slika 6).
Interaktivni proteini
urediDvije interakcije otkrivene putem dva hibridna metoda, dogodile su se sa SSBP3 i ATF4, a obje su faktori transkripcije.
ATF4 / CREB-2 je transkripcijski faktor koji se veže za dugo terminalno ponavljanje virusa ljudske T-ćelijske leukemije tipa1 (HTLV-1). Može biti aktivator HTLV-1.[46][47][48]
SSBP3 / CSDP nalazi se u matičnim ćelijama mišjih embriona, da bi se rzvili u trofoblast (pružaju hranjive sastojke embrionu).[46][49] ZNF226 se eksprimira na većim nivoima unutar ljudskih matičnih ćelija.[24]
Funkcija
urediKako je ZNF226 faktor transkripcije, koji ima ulogu u represiji transkripcije, predviđa se da će se 18 domena vezanja zf-C2H2 vezati za DNK sekvencu prikazanu u logotipu sekvence (slika 7–9).[9][10]
Klinički značaj
urediPovezane bolesti i stanja
urediMutacija unutar gena ZNF226 pozitivno je povezana s prisustvom hepatoćelijskim karcinomom (HCC).[50] Određeni jednostruki nukleotidni polimorfizmi (SNP) (rs2927438) također je u korelaciji sa povećanom ekspresijom ZNF226 u moždanom tkivu čeonog korteksa i perifernim monojedarnim ćelijama, poput T– i B-ćelija.[51] Utvrđeno je da je promotorska regija ZNF226 hipometilirana kod onih koji su bili izloženi kineskoj gladi.[52] Pokazalo se da hipometilirano područje u ZNF226 ima korelaciju metilacije u krvi i prefrontalnom korteksu, iako se ne razumije tačna funkcija proteina u gladi.[52] Gen ZNF226 je naveden među mnogim drugim genima sa varijacijom broja kopija (CNV) koji je povezan sa pojedinačnom zajedničkom varijabilnom imunodeficijencijom (CVID).
SNP-ovi
urediBrojni SNP identificirani su u cijelom genu ZNF226. Unutar promotora GXP_7536741 pronađena su dva SNP-a od interesa. Dolje su navedeni povezani transkripcijski faktori za oba SNP-a. Pokazalo se da hipometilirano područje u ZNF226 ima korelaciju metilacije u krvi i prečeonom korteksu, iako se ne razumije tačna funkcija proteina u gladi.[52] Gen ZNF226 je naveden među mnogim drugim genima sa varijacijom broja kopija (CNV) koji je povezan sa pojedinačnom običnom varijabilnom imunodeficijencijom (CVID).
| Matrica | Puni naziv matrice | Startni položaj | Položaj kraja | Lanac | Matrični rezultat | Funkcija |
| V$LHX4.01 | LIM homeoboks 4, Gsh4 | 58 | 80 | (+) | 0,849 | Homeodomeni imaju ulogu u razvoju kičmenjaka. LIM domeni sadrže dva motiva sa dvostrukim cinkovim prstima i regije bogate cisteinom. Ima ulogu u razvoju nervnih i limfoidnih ćelija.[53][54] |
| V$BRN2.04 | POU klasa 3 homeoboksa 2 (POU3F2), OTF7 | 61 | 79 | (–) | 0,843 | Pokazalo se da POU domen ima ulogu u neuroendokrinom razvoju.[53][55] |
| V$GSH2.01 | Homeodomenski transkripcijski faktor Gsh-2 | 61 | 79 | (+) | 0,966 | Utvrđeno je da Gsh-2 ima ulogu u razvoju mozga.[56] |
| V$LBX2.01 | Bubamarski homeoboks 2 | 61 | 79 | (–) | 0,893 | Pokazano je da kod miševa ovaj TF ima ulogu u sjemenicima i epididimisu.[57] |
| V$CDP.02 | Transcripcijski represor CDP | 149 | 171 | (–) | 0,954 | CDP je proučavan zbog uloge u regulaciji S-faze ćelijskog ciklusa.[58] |
| V$HOXC13.01 | Homeodomenski transcripcijski faktor HOXC13 | 154 | 170 | (–) | 0,917 | HOXC13 ima ulogu u regulaciji ekspresije keratina.[59] |
| V$NFY.04 | Jedarni faktor Y (vezujući faktor Y-boksa) | 155 | 169 | (–) | 0,938 | Ima ulogu u regulaciji ćelijskogog ciklusa i veže se za boks CCAAT uzvodno od mjesta transkripcije.[60] |
Reference
uredi- ^ a b c GRCh38: Ensembl release 89: ENSG00000167380 - Ensembl, maj 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000087598 - Ensembl, maj 2017
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ a b c d e f g h i j k "Entrez Gene: Zinc finger protein 226". Pristupljeno 28. 1. 2016.
- ^ "GeneCards entry on ZNF226". 2020.
- ^ a b c "ZNF226 zinc finger protein 226 [Homo sapiens (human)] - Gene - NCBI". www.ncbi.nlm.nih.gov. Pristupljeno 20. 10. 2020.
- ^ a b Shannon M, Hamilton AT, Gordon L, Branscomb E, Stubbs L (juni 2003). "Differential expansion of zinc-finger transcription factor loci in homologous human and mouse gene clusters". Genome Research. 13 (6A): 1097–110. doi:10.1101/gr.963903. PMC 403638. PMID 12743021.
- ^ a b Persikov AV, Rowland EF, Oakes BL, Singh M, Noyes MB (februar 2014). "Deep sequencing of large library selections allows computational discovery of diverse sets of zinc fingers that bind common targets". Nucleic Acids Research. 42 (3): 1497–508. doi:10.1093/nar/gkt1034. PMC 3919609. PMID 24214968.
- ^ a b Persikov AV, Wetzel JL, Rowland EF, Oakes BL, Xu DJ, Singh M, Noyes MB (februar 2015). "A systematic survey of the Cys2His2 zinc finger DNA-binding landscape". Nucleic Acids Research. 43 (3): 1965–84. doi:10.1093/nar/gku1395. PMC 4330361. PMID 25593323.
- ^ a b "Expasy Compute pI/MW tool".
- ^ a b c Madeira F, Park YM, Lee J, Buso N, Gur T, Madhusoodanan N, et al. (juli 2019). "The EMBL-EBI search and sequence analysis tools APIs in 2019". Nucleic Acids Research. 47 (W1): W636–W641. doi:10.1093/nar/gkz268. PMC 6602479. PMID 30976793.
- ^ Combet C, Blanchet C, Geourjon C, Deléage G (mart 2000). "NPS@: network protein sequence analysis". Trends in Biochemical Sciences. 25 (3): 147–50. doi:10.1016/s0968-0004(99)01540-6. PMID 10694887.
- ^ Yang J, Zhang Y (juli 2015). "I-TASSER server: new development for protein structure and function predictions". Nucleic Acids Research. 43 (W1): W174-81. doi:10.1093/nar/gkv342. PMC 4489253. PMID 25883148.
- ^ Zhang C, Freddolino PL, Zhang Y (juli 2017). "COFACTOR: improved protein function prediction by combining structure, sequence and protein-protein interaction information". Nucleic Acids Research. 45 (W1): W291–W299. doi:10.1093/nar/gkx366. PMC 5793808. PMID 28472402.
- ^ a b "Genomatix Website". Arhivirano s originala, 2. 6. 2021.
- ^ Ray-Gallet D, Mao C, Tavitian A, Moreau-Gachelin F (juli 1995). "DNA binding specificities of Spi-1/PU.1 and Spi-B transcription factors and identification of a Spi-1/Spi-B binding site in the c-fes/c-fps promoter". Oncogene. 11 (2): 303–13. PMID 7624145.
- ^ Horvath CM, Wen Z, Darnell JE (april 1995). "A STAT protein domain that determines DNA sequence recognition suggests a novel DNA-binding domain". Genes & Development. 9 (8): 984–94. doi:10.1101/gad.9.8.984. PMID 7774815.
- ^ Dejosez M, Levine SS, Frampton GM, Whyte WA, Stratton SA, Barton MC, et al. (juli 2010). "Ronin/Hcf-1 binds to a hyperconserved enhancer element and regulates genes involved in the growth of embryonic stem cells". Genes & Development. 24 (14): 1479–84. doi:10.1101/gad.1935210. PMC 2904937. PMID 20581084.
- ^ Otaki M, Hatano M, Kobayashi K, Ogasawara T, Kuriyama T, Tokuhisa T (septembar 2000). "Cell cycle-dependent regulation of TIAP/m-survivin expression". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1493 (1–2): 188–94. doi:10.1016/S0167-4781(00)00142-1. PMID 10978521.
- ^ Dezso Z, Nikolsky Y, Sviridov E, Shi W, Serebriyskaya T, Dosymbekov D, et al. (novembar 2008). "A comprehensive functional analysis of tissue specificity of human gene expression". BMC Biology. 6 (1): 49. doi:10.1186/1741-7007-6-49. PMC 2645369. PMID 19014478.
- ^ Kaur S, Archer KJ, Devi MG, Kriplani A, Strauss JF, Singh R (oktobar 2012). "Differential gene expression in granulosa cells from polycystic ovary syndrome patients with and without insulin resistance: identification of susceptibility gene sets through network analysis". The Journal of Clinical Endocrinology and Metabolism. 97 (10): E2016-21. doi:10.1210/jc.2011-3441. PMC 3674289. PMID 22904171.
- ^ "GEO Profiles entry on ZNF226 Papillary Thyroid Cancer".
- ^ a b c d Cheng CC, Lo HH, Huang TS, Cheng YC, Chang ST, Chang SJ, Wang HW (septembar 2012). "Genetic module and miRNome trait analyses reflect the distinct biological features of endothelial progenitor cells from different anatomic locations". BMC Genomics. 13 (1): 447. doi:10.1186/1471-2164-13-447. PMC 3443421. PMID 22943456.
- ^ a b Persson H, Kvist A, Rego N, Staaf J, Vallon-Christersson J, Luts L, et al. (januar 2011). "Identification of new microRNAs in paired normal and tumor breast tissue suggests a dual role for the ERBB2/Her2 gene". Cancer Research. 71 (1): 78–86. doi:10.1158/0008-5472.CAN-10-1869. PMID 21199797.
- ^ Wang HJ, Zhang PJ, Chen WJ, Jie D, Dan F, Jia YH, Xie LX (juni 2013). "Characterization and Identification of novel serum microRNAs in sepsis patients with different outcomes". Shock. 39 (6): 480–7. doi:10.1097/SHK.0b013e3182940cb8. PMID 23612084. S2CID 43871162.
- ^ Hu SL, Cui GL, Huang J, Jiang JG, Wang DW (septembar 2016). "An APOC3 3'UTR variant associated with plasma triglycerides levels and coronary heart disease by creating a functional miR-4271 binding site". Scientific Reports. 6 (1): 32700. doi:10.1038/srep32700. PMC 5021972. PMID 27624799.
- ^ Ladewig E, Okamura K, Flynt AS, Westholm JO, Lai EC (septembar 2012). "Discovery of hundreds of mirtrons in mouse and human small RNA data". Genome Research. 22 (9): 1634–45. doi:10.1101/gr.133553.111. PMC 3431481. PMID 22955976.
- ^ Frazier S, McBride MW, Mulvana H, Graham D (april 2020). "From animal models to patients: the role of placental microRNAs, miR-210, miR-126, and miR-148a/152 in preeclampsia". Clinical Science. 134 (8): 1001–1025. doi:10.1042/CS20200023. PMC 7239341. PMID 32337535.
- ^ Fong LY, Taccioli C, Palamarchuk A, Tagliazucchi GM, Jing R, Smalley KJ, et al. (mart 2020). "Abrogation of esophageal carcinoma development in miR-31 knockout rats". Proceedings of the National Academy of Sciences of the United States of America. 117 (11): 6075–6085. doi:10.1073/pnas.1920333117. PMC 7084137. PMID 32123074.
- ^ a b Wang WH, Chen J, Zhao F, Zhang BR, Yu HS, Jin HY, Dai JH (2014). "MiR-150-5p suppresses colorectal cancer cell migration and invasion through targeting MUC4". Asian Pacific Journal of Cancer Prevention. 15 (15): 6269–73. doi:10.7314/APJCP.2014.15.15.6269. PMID 25124610.
- ^ "GeneCards entry on FUS".
- ^ "GeneCards entry on RBMY1A1".
- ^ a b "GeneCards entry on RBMX".
- ^ a b "GeneCards entry on ACO1".
- ^ "Expasy Myristoylator tool".
- ^ Xue Y, Liu Z, Cao J, Ma Q, Gao X, Wang Q, et al. (mart 2011). "GPS 2.1: enhanced prediction of kinase-specific phosphorylation sites with an algorithm of motif length selection". Protein Engineering, Design & Selection. 24 (3): 255–60. doi:10.1093/protein/gzq094. PMID 21062758.
- ^ Xue Y, Ren J, Gao X, Jin C, Wen L, Yao X (septembar 2008). "GPS 2.0, a tool to predict kinase-specific phosphorylation sites in hierarchy". Molecular & Cellular Proteomics. 7 (9): 1598–608. doi:10.1074/mcp.M700574-MCP200. PMC 2528073. PMID 18463090.
- ^ Xue Y, Zhou F, Zhu M, Ahmed K, Chen G, Yao X (juli 2005). "GPS: a comprehensive www server for phosphorylation sites prediction". Nucleic Acids Research. 33 (Web Server issue): W184-7. doi:10.1093/nar/gki393. PMC 1160154. PMID 15980451.
- ^ "Motif Scan tool".
- ^ a b Cui B, Pan Q, Clarke D, Villarreal MO, Umbreen S, Yuan B, et al. (oktobar 2018). "S-nitrosylation of the zinc finger protein SRG1 regulates plant immunity". Nature Communications. 9 (1): 4226. Bibcode:2018NatCo...9.4226C. doi:10.1038/s41467-018-06578-3. PMC 6185907. PMID 30315167.
- ^ Guidez F, Howell L, Isalan M, Cebrat M, Alani RM, Ivins S, et al. (juli 2005). "Histone acetyltransferase activity of p300 is required for transcriptional repression by the promyelocytic leukemia zinc finger protein". Molecular and Cellular Biology. 25 (13): 5552–66. doi:10.1128/MCB.25.13.5552-5566.2005. PMC 1156991. PMID 15964811.
- ^ Jen J, Wang YC (juli 2016). "Zinc finger proteins in cancer progression". Journal of Biomedical Science. 23 (1): 53. doi:10.1186/s12929-016-0269-9. PMC 4944467. PMID 27411336.
- ^ Wei HM, Hu HH, Chang GY, Lee YJ, Li YC, Chang HH, Li C (maj 2014). "Arginine methylation of the cellular nucleic acid binding protein does not affect its subcellular localization but impedes RNA binding". FEBS Letters. 588 (9): 1542–8. doi:10.1016/j.febslet.2014.03.052. PMID 24726729. S2CID 207645796.
- ^ a b "PSORT II tool".
- ^ a b Ravasi T, Suzuki H, Cannistraci CV, Katayama S, Bajic VB, Tan K, et al. (mart 2010). "An atlas of combinatorial transcriptional regulation in mouse and man". Cell. 140 (5): 744–52. doi:10.1016/j.cell.2010.01.044. PMC 2836267. PMID 20211142.
- ^ "NCBI entry on ATF4".
- ^ Gachon F, Peleraux A, Thebault S, Dick J, Lemasson I, Devaux C, Mesnard JM (oktobar 1998). "CREB-2, a cellular CRE-dependent transcription repressor, functions in association with Tax as an activator of the human T-cell leukemia virus type 1 promoter". Journal of Virology. 72 (10): 8332–7. doi:10.1128/JVI.72.10.8332-8337.1998. PMC 110203. PMID 9733879.
- ^ Liu J, Luo X, Xu Y, Gu J, Tang F, Jin Y, Li H (maj 2016). "Single-stranded DNA binding protein Ssbp3 induces differentiation of mouse embryonic stem cells into trophoblast-like cells". Stem Cell Research & Therapy. 7 (1): 79. doi:10.1186/s13287-016-0340-1. PMC 4884356. PMID 27236334.
- ^ Fujimoto A, Totoki Y, Abe T, Boroevich KA, Hosoda F, Nguyen HH, et al. (maj 2012). "Whole-genome sequencing of liver cancers identifies etiological influences on mutation patterns and recurrent mutations in chromatin regulators". Nature Genetics. 44 (7): 760–4. doi:10.1038/ng.2291. PMID 22634756. S2CID 54585617.
- ^ Rao S, Ghani M, Guo Z, Deming Y, Wang K, Sims R, et al. (juni 2018). "An APOE-independent cis-eSNP on chromosome 19q13.32 influences tau levels and late-onset Alzheimer's disease risk". Neurobiology of Aging. 66: 178.e1–178.e8. doi:10.1016/j.neurobiolaging.2017.12.027. PMC 7050280. PMID 29395286.
- ^ a b c He Y, de Witte LD, Houtepen LC, Nispeling DM, Xu Z, Yu Q, et al. (maj 2019). "DNA methylation changes related to nutritional deprivation: a genome-wide analysis of population and in vitro data". Clinical Epigenetics. 11 (1): 80. doi:10.1186/s13148-019-0680-7. PMC 6524251. PMID 31097004.
- ^ a b Berger MF, Badis G, Gehrke AR, Talukder S, Philippakis AA, Peña-Castillo L, et al. (juni 2008). "Variation in homeodomain DNA binding revealed by high-resolution analysis of sequence preferences". Cell. 133 (7): 1266–76. doi:10.1016/j.cell.2008.05.024. PMC 2531161. PMID 18585359.
- ^ Wang X, He C, Hu X (maj 2014). "LIM homeobox transcription factors, a novel subfamily which plays an important role in cancer (review)". Oncology Reports. 31 (5): 1975–85. doi:10.3892/or.2014.3112. PMID 24676471.
- ^ Assa-Munt N, Mortishire-Smith RJ, Aurora R, Herr W, Wright PE (april 1993). "The solution structure of the Oct-1 POU-specific domain reveals a striking similarity to the bacteriophage lambda repressor DNA-binding domain". Cell. 73 (1): 193–205. doi:10.1016/0092-8674(93)90171-l. PMID 8462099. S2CID 24276357.
- ^ Hsieh-Li HM, Witte DP, Szucsik JC, Weinstein M, Li H, Potter SS (april 1995). "Gsh-2, a murine homeobox gene expressed in the developing brain". Mechanisms of Development. 50 (2–3): 177–86. doi:10.1016/0925-4773(94)00334-j. PMID 7619729. S2CID 14104595.
- ^ Moisan V, Bomgardner D, Tremblay JJ (februar 2008). "Expression of the Ladybird-like homeobox 2 transcription factor in the developing mouse testis and epididymis". BMC Developmental Biology. 8 (1): 22. doi:10.1186/1471-213X-8-22. PMC 2277406. PMID 18304314.
- ^ Truscott M, Raynal L, Premdas P, Goulet B, Leduy L, Bérubé G, Nepveu A (april 2003). "CDP/Cux stimulates transcription from the DNA polymerase alpha gene promoter". Molecular and Cellular Biology. 23 (8): 3013–28. doi:10.1128/MCB.23.8.3013-3028.2003. PMC 152546. PMID 12665598.
- ^ Jave-Suarez LF, Winter H, Langbein L, Rogers MA, Schweizer J (februar 2002). "HOXC13 is involved in the regulation of human hair keratin gene expression". The Journal of Biological Chemistry. 277 (5): 3718–26. doi:10.1074/jbc.M101616200. PMID 11714694.
- ^ Zhao H, Wu D, Kong F, Lin K, Zhang H, Li G (2017). "Arabidopsis thaliana Nuclear Factor Y Transcription Factors". Frontiers in Plant Science (jezik: English). 7: 2045. doi:10.3389/fpls.2016.02045. PMC 5222873. PMID 28119722.CS1 održavanje: nepoznati jezik (link)



