RNK interferencija
Interferencija RNK (RNKi) je biološki proces u kojem su RNK molekule uključene u supresiju ekspresije gena specifičnu za sekvencu dvolančane RNK, putem translacijske ili transkripcijske represije . Historijski gledano, RNKi je bila poznata pod drugim nazivima, uključujući "kosupresiju", "utišavanje gena nakon transkripcije" (PTGS) i "gušenje". Detaljno proučavanje svakog od ovih naizgled različitih procesa razjasnilo je da su svi identiteti ovih fenomena zapravo RNKi. Andrew Fire i Craig C. Mello podijelili su 2006. Nobelovu nagradu za fiziologiju ili medicinu za svoj rad na RNKi u obloj glisti Caenorhabditis elegans, koji su objavili 1998. Od otkrića RNKi i njegovih regulatornih potencijala, postalo je očigledno da RNKi ima ogroman potencijal u supresiji željenih gena. RNKi je sada poznata kao precizan, efikasan, stabilan i supresor, bolji od antisens terapija za supresiju gena.[1] Antisens RNK proizvedena unutar ćelija, ekspresijskim vektorom može se razviti i naći upotrebnu kao novi terapeutski agens.[2]
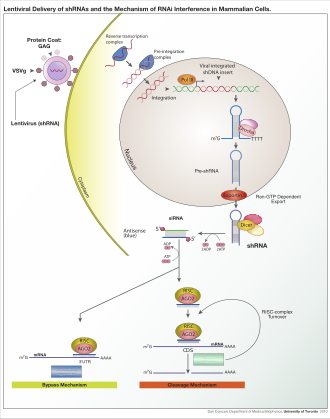
Dva tipa malih molekula RNK (RNK) – mikroRNK (miRNK) i mala interferirajuća RNK (siRNK) – centralne su za interferenciju RNK. RNK su direktni proizvodi gena, a ove male RNK mogu usmjeravati enzimske komplekse da razgrađuju molekule informacijske RNK (iRNK) i na taj način smanjuju njihovu aktivnost sprečavanjem translacije, putem utišavanja gena nakon transkripcije. Štaviše, transkripcija se može inhibirati putem mehanizma za utišavanje prije transkripcije interferencije RNK, kroz koji enzimski kompleks katalizira metilaciju DNK na genomskim pozicijama komplementarnim kompleksiranoj siRNK ili miRNK. Interferencija RNK ima važnu ulogu u odbrani ćelija od parazitskih nukleotid nihsekvenci – virusa i transpozona. Također utiče na razvoj.
RNKi put nalazi se kod mnogih eukariota, uključujući životinje, a pokreće ga enzim Dicer, koji cijepa molekule duge dvolančane RNK (dsRNK) na kratke dvolančane fragmente od ~21 nukleotida siRNK. Svaka siRNA odmotava se u dvije jednolančane RNK (ssRNK), putnički lanac i lanac vodiča. Putnički lanac je degradiran, a lanac-vodič ugrađen je u RNK-inducirani kompleks za utišavanje (RISC). Najbolje proučavani ishod je utišavanje gena nakon transkripcije, koje se događa kada se lanac vodič upari s komplementarnom sekvencom u molekuli iRNK i inducira cijepanje pomoću argonaut 2 (Ago2), katalitske komponente RISC-a. U nekim organizmima, ovaj proces se sistemski širi, uprkos početno ograničenim molarnim koncentracijama siRNK.
RNKi je vrijedan istraživački alat, kako u kulturi ćelija tako iu živim organizmima, jer sintetska dsRNK uvedena u ćelije može selektivno i snažno inducirati supresiju specifičnih gena od interesa. RNKi može se koristiti za velike skrininge koji sistematski isključuju svaki gen u ćeliji, što može pomoći da se identifikuju komponente neophodne za određeni ćelijski proces ili događaj kao što je ćelijska dioba. Put se također koristi kao praktičan alat u biotehnologijama, medicini i proizvodnji insekticida.[3]
Ćelijski mehanizam
urediRNKi je proces RNK-ovisnog utišavanja gena koji je kontroliran RNK-induciranim kompleksom utišavanja (RISC) i iniciran je kratkim molekulama dvolančane RNK u citoplazmi ćelije, gdje stupaju u interakciju sa katalitskom RISC komponentom argonauta.[5] Kada je dsRNK egzogena (dolazi od infekcije virusom sa RNA genomom ili laboratorijskim manipulacijama), RNK se uvozi direktno u citoplazmu i Dicer cijepa na kratke fragmente. Početna dsRNK također može biti endogena (porijeklom iz ćelije), kao u premikroRNK eksprimiranim iz RNK-nkodirajućih gena u genomu. Primarni transkripti iz takvih gena se prvo obrađuju da formiraju karakterističnu strukturu matične petlje pre-miRNK u nukleus, a zatim se eksportuju u citoplazmu. Dakle, dva puta dsRNK, egzogena i endogena, konvergiraju na RISC.[6]
Egzogena dsRNK inicira RNAi aktiviranjem ribonukleaznog proteina Dicer,[7] koji veže se i cijepa dvolančane RNK (dsRNK) u biljkama ili kratke RNK ukosnice (shRNK) kod ljudi, kako bi se proizveli dvolančani fragmenti od 20–25 baznih parova s prekoračenjem od dva nukleotida na 3′ kraju.[8] Bioinformatičke studije o genomima više organizama sugeriraju da ova dužina maksimizira specifičnost ciljanog gena i minimizira nespecifične efekte.[9] Ovi kratki dvolančani fragmenti nazivaju se male interferirajuće RNK (siRNK). Ove siRNK razdvajaju se zatim u pojedinačne lančiće i integriraju u aktivni RISC, pomoću RISC-učitavajućeg kompleksa (RLC). RLC uključuje Dicer-2 i R2D2, a ključan je za ujedinjavanje Ago2 i RISC-a.[10] TATA-vezujući proteinski vezani faktor 11 (TAF11) sastavlja RLC, olakšavajući tetramerizaciju Dcr-2-R2D2, što povećava afinitet vezivanja za siRNA za 10 puta. Povezivanje sa TAF11 bi pretvorilo kompleks R2-D2-inicijatora (RDI) u RLC.[11] R2D2 nosi tandem dvolančane RNK-vezujućeg domena za prepoznavanje termodinamički stabilnog kraja dupleksa siRNK, dok je Dicer-2 drugi manje stabilan ekstremitet. Učitavanje je asimetrično: MID domen Ago2 prepoznaje termodinamički stabilan kraj siRNK. Zbog toga se se izbacuje "putnički" (sens) lanac čiji 5′ kraj odbacuje MID, dok sačuvani "vodič" (antisens) lanac sarađuje sa AGO da bi formirao RISC.[10]
Nakon integracije u RISC, siRNK bazni par sa svojom ciljnom iRNK i cijepa je, čime se sprečava da se koristi kao translacijski šablon.[12] Za razliku od siRNK, miRNK napunjen RISC kompleks skenira citoplazmatsku iRNK za potencijalnu komplementarnost. Umjesto destruktivnog cijepanja (putem Ago2), miRNK radije ciljaju na 3' UTR regione iRNK, gdje se obično vezuju nesavršenom komplementarnošću, blokirajući tako pristup ribosomima za translaciju.[13]
Egzogenu dsRNK detektuje i vezuje efektorski protein, poznat kao RDE-4 u C. elegans i R2D2 u Drosophila, koji stimulira aktivnost dicera.[14] Mehanizam koji proizvodi ovu specifičnost dužine je nepoznat i ovaj protein vezuje samo dugačke dsRNK.[14]
U C. elegans ovaj inicijacijski odgovor pojačava se sintezom molekukla 'sekundarne' siRNK tokom koje se inicirajuće ili 'primarne' siRNK koriste kao šabloni.[15] Ove 'sekundarne' siRNK razlikuju se od strukturno siRNK i proizvodi ih RNK-ovisna RNK-polimeraza (RdRP).[16][17]
MikroRNK
urediMikroRNK (miRNA) su genomski kodirane nekodirajuće RNK koje pomažu u regulaciji ekspresije gena, posebno tokom razvoja.[18] Fenomen RNKi, široko definisan, uključuje endogeno indukovane efekte utišavanja gena miRNK, kao i utišavanje izazvano stranim dsRNK. Zrele miRNK su strukturno slične siRNK proizvedene od egzogene dsRNK, ali prije nego što dostignu zrelost, miRNK moraju prvo proći opsežnu posttranskripcijsku modifikaciju. MiRNK eksprimira se iz mnogo dužeg gena koji kodira RNK kao primarni transkript, poznat kao "pri-miRNK" koji se prerađuje u ćelijskom jedru, u 70-nukleotidnoj strukturi matične petlje koja se naziva "pre-miRNK". Ovaj kompleks sastoji se od enzima RNaza III zvanog Drosha i proteina koji vezuje dsRNK DGCR8. Dicer dsRNA ove pre-miRNK vezuje i cijepa Dicer da bi proizveo zrelu miRNK molekulu koja se može integrirati u kompleks RISC-a; tako, miRNK i siRNK dijele isti ćelijski nizvodni mehanizam.[19] Prve, virusno [[kodon|kodirane miRNK opisane su kod Epstein-Barrovog virusa (EBV).[20] Nakon toga, u virusima opisivan je sve veći broj mikroRNK. VIRmiRNK je sveobuhvatan katalog koji pokriva virusne mikroRNK, njihove mete i antivirusne miRNK.[21][22] siRNK izvedene iz dugih prekursora dsRNK razlikuju se od miRNK po tome što miRNK, posebno one kod životinja, obično imaju nepotpuno uparivanje baza sa metom i inhibiraju translaciju mnogih različitih iRNK sa sličnim sekvencama. Nasuprot tome, siRNK obično se savršeno uparuju bazama i induciraju cijepanje iRNK samo u jednoj, specifičnoj meti.[23] U Drosophila melanogaster i C. elegans, miRNK i siRNK obrađuju se različitim proteinima argonauta i dicer enzimima.[24][25]
Tri primarne neprevedene regije i mikroRNK
urediTri primarne neprevedene regije (3′UTR) iRNK često sadrže regulatorne sekvence koje nakon transkripcije uzrokuju RNKi. Takvi 3′-UTR često sadrže i mjesta vezanja za mikroRNK (miRNK) kao i za regulatorne proteine. Vezivanjem za specifična mjesta unutar 3′-UTR, miRNK mogu smanjiti ekspresiju gena različitih iRNK, tako što inhibiraju translaciju ili direktno uzrokuju degradaciju transkripta. 3' UTR također može imati prigušivače regija koje vezuju represorske proteine koji inhibiraju ekspresiju iRNK.
3'-UTR često sadrži mikroRNK elemente odgovora (MRE). MRE su sekvence za koje se vežu miRNK. Ovo su preovlađujući motivi unutar 3′-UTR-a. Među svim regulatornim motivima unutar 3′-UTR (npr. uključujući regije prigušivača), MRE čine otprilike polovinu motiva.
Od 2014. godine, miRBase web stranica,[26] arhiva miRNK [sekvenci i napomena, navela je 28.645 unosa u 233 biološke vrste. Od toga, 1.881 miRNK bila je u označenim ljudskim lokusima miRNK. Predviđeno je da miRNK imaju u prosjeku oko četiri stotine ciljnih iRNK (što utiče na ekspresiju nekoliko stotina gena).[27] Friedman et al.[27] procjenjuju se da je >45.000 ciljnih mjesta miRNK unutar 3' UTR ljudske iRNK konzervirano iznad pozadinskih nivoa, a >60% gena koji kodiraju ljudske proteine je pod selekcijskim pritiskom da održi uparivanje s miRNK.
Direktni eksperimenti pokazuju da jedna miRNK može smanjiti stabilnost stotina jedinstvenih iRNK.[28] Drugi eksperimenti pokazuju da jedna miRNK može potisnuti proizvodnju stotina proteina, ali da je ta represija često relativno blaža (manje od dva puta).[29][30]
Efekti disregulacije ekspresije gena miRNK važni kod kancera.[31] Naprimjer, kod karcinoma gastrointestinalnog trakta, devet miRNK je identificirano kao epigenetički izmijenjenih i efikasnih u smanjenju regulacije enzima za popravak DNK.[32]
Efekti disregulacije ekspresije gena miRNK važni su i kod neuropsihijatrijskih poremećaja, kao što su shizofrenija, bipolarni poremećaj, velika depresija, Parkinsonova bolest, Alzheimerova bolest i poremećaji iz autističnog spektra.[33][34][35]
RISC aktivacija i kataliza
urediEgzogenu dsRNK detektuje i vezuje efektorski protein, poznat kao RDE-4 u Caenorhabditis elegans i R2D2 u Drosophila, koji stimuliše aktivnost dicera.[14] Ovaj protein veže samo dugačke dsRNK, ali mehanizam koji proizvodi ovu specifičnost dužine je nepoznat.[14] Ovaj protein koji se vezuje za RNK zatim olakšava prijenos cijepanih siRNA u kompleks RISC.[36]
U C. elegans ovaj inicijacijski odgovor se pojačava kroz sintezu populacije 'sekundarne' siRNK tokom koje se inicirajuće ili 'primarne' siRNK koriste kao predlošci.[15] Ove 'sekundarne' siRNK su strukturno različite od dicerski proizvedenih siRNK i izgleda da su proizvedene RNK-ovisna RNK-polimeraza (RdRP).[16][17]
Desno: PIWI domen proteina argonaut u kompleksu sa dvolančanom RNK.
Aktivne komponente RNK-indukovanog kompleksa utišavanja (RISC) su endonukleaze zvane argonautski proteini, koji cijepaju ciljni mRNK lanac komplementarno na njihovu vezu s siRNA.[5] Kako su fragmenti koje proizvodi dicer dvolančani, svaki bi u teoriji mogao proizvesti funkcionalnu siRNA. Međutim, samo jedan od dva lanca, koji je poznat kao "vodič", vezuje protein argonauta i usmjerava utišavanje gena. Drugi "anti-vodič" ili "putnička sekvenca" degradiran je tokom RISC aktivacije.[37] Iako se prvo vjerovalo da ATP zavisna helikaza razdvaja ova dva lanca,[38] proces se pokazao kao nezavisan od ATP-a i izveden direktno od proteinskih komponenti RISC-a.[39][40] Međutim, "in vitro" kinetička analiza RNKi u prisustvu i odsustvu ATP-a pokazala je da ATP može biti potreban za odmotavanje i uklanjanje cijepanog lanca mRNK iz RISC kompleksa nakon katalize.[41] Vodički lanac teži da bude onaj čiji je 5' kraj manje stabilno uparen sa svojim komplementom,[42] ali selekcija lanca nije pod utjecajem smjera u kojem dicer cijepa dsRNK prije inkorporacije RISC-a.[43] Umjesto toga, protein R2D2 može poslužiti kao faktor diferencijacije vezivanjem stabilnijeg 5′ kraja putničkog lanca.[44]
Strukturna osnova za vezivanje RNK za protein argonauta ispitana je rendgenskom kristalografijom vezivanja domena proteina argonauta vezanog za RNK. Ovdje fosforilirani 5′ kraj RNK lanca ulazi u konzervirana osnovne površinske džepove i uspostavlja kontakte kroz dvovalentni kation (atom sa dva pozitivna naboja) kao što je magnezij i aromatičnost slaganje (proces koji omogućava više od jednog atom da dijeli elektron prenoseći ga naprijed-nazad) između 5' nukleotida u siRNK i konzerviranog tirozinskog ostatka. Smatra se da ovo mjesto formira mjesto nukleacije za vezivanje siRNK za njenu ciljni iRNK.[45][46] Indeed, RNAi may be more effective against mRNA targets that are not translated.[47] Argonautski proteini lokalizirani su na specifične regije u citoplazmi koje se nazivaju P-tijela (takođe citoplazmatska tijela ili GW tijela), a to su regije s visokim stopama raspada iRNK;[48] Aktivnost miRNK je takođe grupisana u P-tijelima.[49] Poremećaj P-tijela smanjuje efikasnost RNAi, sugerirajući da su ona kritično mjesto u preradi RNKi.[50]
Transkripcijsko utišavanje
urediKomponente RNKi puta koriste se kod mnogih eukariota u održavanju organizacije i strukture njihovih genoma. Modifikacija histona i povezana indukcija formiranja heterohromatina služe za pretranskripcijsko utišavanje gena;[52] ovaj proces se naziva RNK-inducirano utišavanje transkripcije (RITS), a provodi ga kompleks proteina koji se naziva RITS kompleks. U fisijskom kvascu ovaj kompleks sadrži argonaut, hromodomenski protein Chp1 i protein zvanu Tas3 nepoznate funkcije.[53] Kao posljedica toga, indukcija i širenje heterokromatskih regija zahtijeva argonautske i RdRP proteine.[54] Zaista, delecija ovih gena u fisijskom kvascu S. pombe ometa metilaciju histona i formiranje centromera,[55] uzrokujući usporenu ili zaustavljenu anafazu tokom ćelijskih dioba.[56] U nekim slučajevima, uočeni su slični procesi povezani sa modifikacijom histona za transkripcijsku regulaciju gena.[57]
Mehanizam kojim RITS kompleks indukuje formiranje i organizaciju heterohromatina nije dobro shvaćen. Većina studija fokusirala se na regiju tipa parenja u fisijskom kvascu, koji možda nije reprezentativan za aktivnosti u drugim genomskim regijama/organizmima. U održavanju postojećih heterokromatinskih regija, RITS formira kompleks sa siRNK komplementaran za lokalne gene i stabilno vezuje lokalne metilirane histone, djelujući kotranskripcijski, kako bi razgradili sve novonastale pre-iRNK transkripte koji su inicirani RNK-polimerazama. Formiranje takvog heterohromatinskog regiona, iako ne njegovo održavanje, zavisi od dicer-a, vjerovatno zato što je dicer potreban da generira početni komplement siRNK koji cilja naknadne transkripte.[58] Sugerirano je da održavanje heterohromatina funkcionira kao povratna petlja koja se samopojačava, jer se nove siRNK formiraju iz povremenih transkripata u nastajanju putem RdRP-a za ugradnju u lokalne RITS komplekse.[59] Relevantnost zapažanja iz regija fisijskog kvasca koji se pare i sisarskih centromera nije jasna, jer održavanje heterohromatina u ćelijama sisara može biti nezavisno od komponenti RNKi puta.[60]
Također pogledajte
urediReferences
uredi- ^ Saurabh S, Vidyarthi AS, Prasad D (mart 2014). "RNA interference: concept to reality in crop improvement". Planta. 239 (3): 543–64. doi:10.1007/s00425-013-2019-5. PMID 24402564.
- ^ Weiss B, Davidkova G, Zhou LW (mart 1999). "Antisense RNA gene therapy for studying and modulating biological processes". Cellular and Molecular Life Sciences. 55 (3): 334–58. doi:10.1007/s000180050296. PMID 10228554. S2CID 9448271.
- ^ Kupferschmidt K (august 2013). "A lethal dose of RNA". Science. 341 (6147): 732–3. Bibcode:2013Sci...341..732K. doi:10.1126/science.341.6147.732. PMID 23950525.
- ^ Macrae IJ, Zhou K, Li F, Repic A, Brooks AN, Cande WZ, Adams PD, Doudna JA (januar 2006). "Structural basis for double-stranded RNA processing by Dicer". Science. 311 (5758): 195–8. Bibcode:2006Sci...311..195M. doi:10.1126/science.1121638. PMID 16410517. S2CID 23785494.
- ^ a b Daneholt B. "Advanced Information: RNA interference". The Nobel Prize in Physiology or Medicine 2006. Arhivirano s originala, 20. 1. 2007. Pristupljeno 25. 1. 2007.
- ^ Bagasra O, Prilliman KR (august 2004). "RNA interference: the molecular immune system". Journal of Molecular Histology. 35 (6): 545–53. CiteSeerX 10.1.1.456.1701. doi:10.1007/s10735-004-2192-8. PMID 15614608. S2CID 2966105.
- ^ Bernstein E, Caudy AA, Hammond SM, Hannon GJ (januar 2001). "Role for a bidentate ribonuclease in the initiation step of RNA interference". Nature. 409 (6818): 363–6. Bibcode:2001Natur.409..363B. doi:10.1038/35053110. PMID 11201747. S2CID 4371481.
- ^ Siomi H, Siomi MC (januar 2009). "On the road to reading the RNA-interference code". Nature. 457 (7228): 396–404. Bibcode:2009Natur.457..396S. doi:10.1038/nature07754. PMID 19158785. S2CID 205215974.
Zamore PD, Tuschl T, Sharp PA, Bartel DP (mart 2000). "RNAi: double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals". Cell. 101 (1): 25–33. doi:10.1016/S0092-8674(00)80620-0. PMID 10778853.
Vermeulen A, Behlen L, Reynolds A, Wolfson A, Marshall WS, Karpilow J, Khvorova A (maj 2005). "The contributions of dsRNA structure to Dicer specificity and efficiency". RNA. 11 (5): 674–82. doi:10.1261/rna.7272305. PMC 1370754. PMID 15811921.
Castanotto D, Rossi JJ (januar 2009). "The promises and pitfalls of RNA-interference-based therapeutics". Nature. 457 (7228): 426–33. Bibcode:2009Natur.457..426C. doi:10.1038/nature07758. PMC 2702667. PMID 19158789. - ^ Qiu S, Adema CM, Lane T (2005). "A computational study of off-target effects of RNA interference". Nucleic Acids Research. 33 (6): 1834–47. doi:10.1093/nar/gki324. PMC 1072799. PMID 15800213.
- ^ a b Nakanishi K (septembar 2016). "Anatomy of RISC: how do small RNAs and chaperones activate Argonaute proteins?". Wiley Interdisciplinary Reviews: RNA. 7 (5): 637–60. doi:10.1002/wrna.1356. PMC 5084781. PMID 27184117.
- ^ Liang C, Wang Y, Murota Y, Liu X, Smith D, Siomi MC, Liu Q (septembar 2015). "TAF11 Assembles the RISC Loading Complex to Enhance RNAi Efficiency". Molecular Cell. 59 (5): 807–18. doi:10.1016/j.molcel.2015.07.006. PMC 4560963. PMID 26257286.
- ^ Ahlquist P (maj 2002). "RNA-dependent RNA polymerases, viruses, and RNA silencing". Science. 296 (5571): 1270–3. Bibcode:2002Sci...296.1270A. doi:10.1126/science.1069132. PMID 12016304. S2CID 42526536.
- ^ Roberts TC (2015). "The microRNA Machinery". Advances in Experimental Medicine and Biology. 887: 15–30. doi:10.1007/978-3-319-22380-3_2. ISBN 978-3-319-22379-7. PMID 26662984.
- ^ a b c d Parker G, Eckert D, Bass B (2006). "RDE-4 preferentially binds long dsRNA and its dimerization is necessary for cleavage of dsRNA to siRNA". RNA. 12 (5): 807–18. doi:10.1261/rna.2338706. PMC 1440910. PMID 16603715.
- ^ a b Baulcombe DC (januar 2007). "Molecular biology. Amplified silencing". Science. 315 (5809): 199–200. doi:10.1126/science.1138030. PMID 17218517. S2CID 46285020.
- ^ a b Pak J, Fire A (januar 2007). "Distinct populations of primary and secondary effectors during RNAi in C. elegans". Science. 315 (5809): 241–4. Bibcode:2007Sci...315..241P. doi:10.1126/science.1132839. PMID 17124291. S2CID 46620298.
- ^ a b Sijen T, Steiner FA, Thijssen KL, Plasterk RH (januar 2007). "Secondary siRNAs result from unprimed RNA synthesis and form a distinct class". Science. 315 (5809): 244–7. Bibcode:2007Sci...315..244S. doi:10.1126/science.1136699. PMID 17158288. S2CID 9483460.
- ^ Wang QL, Li ZH (maj 2007). "The functions of microRNAs in plants". Frontiers in Bioscience. 12: 3975–82. doi:10.2741/2364. PMC 2851543. PMID 17485351. S2CID 23014413.
Zhao Y, Srivastava D (april 2007). "A developmental view of microRNA function". Trends in Biochemical Sciences. 32 (4): 189–97. doi:10.1016/j.tibs.2007.02.006. PMID 17350266. - ^ Gregory RI, Chendrimada TP, Shiekhattar R (2006). "MicroRNA biogenesis: isolation and characterization of the microprocessor complex". MicroRNA Protocols. Methods in Molecular Biology. 342. str. 33–47. doi:10.1385/1-59745-123-1:33. ISBN 978-1-59745-123-9. PMID 16957365.
- ^ Pfeffer S, Zavolan M, Grässer FA, Chien M, Russo JJ, Ju J, John B, Enright AJ, Marks D, Sander C, Tuschl T (april 2004). "Identification of virus-encoded microRNAs". Science. 304 (5671): 734–6. Bibcode:2004Sci...304..734P. doi:10.1126/science.1096781. PMID 15118162. S2CID 25287167.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1. 1. 2014). "VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets". Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ http://crdd.osdd.net/servers/virmirna/).
- ^ Pillai RS, Bhattacharyya SN, Filipowicz W (2007). "Repression of protein synthesis by miRNAs: how many mechanisms?". Trends Cell Biol. 17 (3): 118–26. doi:10.1016/j.tcb.2006.12.007. PMID 17197185.
- ^ Okamura K, Ishizuka A, Siomi H, Siomi M (2004). "Distinct roles for Argonaute proteins in small RNA-directed RNA cleavage pathways". Genes Dev. 18 (14): 1655–66. doi:10.1101/gad.1210204. PMC 478188. PMID 15231716.
- ^ Lee Y, Nakahara K, Pham J, Kim K, He Z, Sontheimer E, Carthew R (2004). "Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways". Cell. 117 (1): 69–81. doi:10.1016/S0092-8674(04)00261-2. PMID 15066283.
- ^ miRBase.org
- ^ a b Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (februar 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (septembar 2008). "Widespread changes in protein synthesis induced by microRNAs". Nature. 455 (7209): 58–63. Bibcode:2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (septembar 2008). "The impact of microRNAs on protein output". Nature. 455 (7209): 64–71. Bibcode:2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, Marques MM (juli 2011). "Mechanisms and role of microRNA deregulation in cancer onset and progression". Genetics and Molecular Biology. 34 (3): 363–70. doi:10.1590/S1415-47572011000300001. PMC 3168173. PMID 21931505.
- ^ Bernstein C, Bernstein H (maj 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ Maffioletti E, Tardito D, Gennarelli M, Bocchio-Chiavetto L (2014). "Micro spies from the brain to the periphery: new clues from studies on microRNAs in neuropsychiatric disorders". Frontiers in Cellular Neuroscience. 8: 75. doi:10.3389/fncel.2014.00075. PMC 3949217. PMID 24653674.
- ^ Mellios N, Sur M (2012). "The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders". Frontiers in Psychiatry. 3: 39. doi:10.3389/fpsyt.2012.00039. PMC 3336189. PMID 22539927.
- ^ Geaghan M, Cairns MJ (august 2015). "MicroRNA and Posttranscriptional Dysregulation in Psychiatry". Biological Psychiatry. 78 (4): 231–9. doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ Liu Q, Rand TA, Kalidas S, Du F, Kim HE, Smith DP, Wang X (septembar 2003). "R2D2, a bridge between the initiation and effector steps of the Drosophila RNAi pathway". Science. 301 (5641): 1921–5. Bibcode:2003Sci...301.1921L. doi:10.1126/science.1088710. PMID 14512631. S2CID 41436233.
- ^ Gregory RI, Chendrimada TP, Cooch N, Shiekhattar R (novembar 2005). "Human RISC couples microRNA biogenesis and posttranscriptional gene silencing". Cell. 123 (4): 631–40. doi:10.1016/j.cell.2005.10.022. PMID 16271387.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology (5th izd.). WH Freeman: New York, NY. ISBN 978-0-7167-4366-8.
- ^ Matranga C, Tomari Y, Shin C, Bartel DP, Zamore PD (novembar 2005). "Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes". Cell. 123 (4): 607–20. doi:10.1016/j.cell.2005.08.044. PMID 16271386.
- ^ Leuschner PJ, Ameres SL, Kueng S, Martinez J (mart 2006). "Cleavage of the siRNA passenger strand during RISC assembly in human cells". EMBO Reports. 7 (3): 314–20. doi:10.1038/sj.embor.7400637. PMC 1456892. PMID 16439995.
- ^ Haley B, Zamore PD (juli 2004). "Kinetic analysis of the RNAi enzyme complex". Nature Structural & Molecular Biology. 11 (7): 599–606. doi:10.1038/nsmb780. PMID 15170178. S2CID 12400060.
- ^ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (oktobar 2003). "Asymmetry in the assembly of the RNAi enzyme complex". Cell. 115 (2): 199–208. doi:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ^ Preall JB, He Z, Gorra JM, Sontheimer EJ (mart 2006). "Short interfering RNA strand selection is independent of dsRNA processing polarity during RNAi in Drosophila". Current Biology. 16 (5): 530–5. doi:10.1016/j.cub.2006.01.061. PMID 16527750.
- ^ Tomari Y, Matranga C, Haley B, Martinez N, Zamore PD (novembar 2004). "A protein sensor for siRNA asymmetry". Science. 306 (5700): 1377–80. Bibcode:2004Sci...306.1377T. doi:10.1126/science.1102755. PMID 15550672. S2CID 31558409.
- ^ Ma JB, Yuan YR, Meister G, Pei Y, Tuschl T, Patel DJ (mart 2005). "Structural basis for 5'-end-specific recognition of guide RNA by the A. fulgidus Piwi protein". Nature. 434 (7033): 666–70. Bibcode:2005Natur.434..666M. doi:10.1038/nature03514. PMC 4694588. PMID 15800629.
- ^ Sen G, Wehrman T, Blau H (2005). "mRNA translation is not a prerequisite for small interfering RNA-mediated mRNA cleavage". Differentiation. 73 (6): 287–93. doi:10.1111/j.1432-0436.2005.00029.x. PMID 16138829. S2CID 41117614.
- ^ Gu S, Rossi J (2005). "Uncoupling of RNAi from active translation in mammalian cells". RNA. 11 (1): 38–44. doi:10.1261/rna.7158605. PMC 1370689. PMID 15574516.
- ^ Sen G, Blau H (2005). "Argonaute 2/RISC resides in sites of mammalian mRNA decay known as cytoplasmic bodies". Nat Cell Biol. 7 (6): 633–6. doi:10.1038/ncb1265. PMID 15908945. S2CID 6085169.
- ^ Lian S, Jakymiw A, Eystathioy T, Hamel J, Fritzler M, Chan E (2006). "GW bodies, microRNAs and the cell cycle". Cell Cycle. 5 (3): 242–5. doi:10.4161/cc.5.3.2410. PMID 16418578.
- ^ Jakymiw A, Lian S, Eystathioy T, Li S, Satoh M, Hamel J, Fritzler M, Chan E (2005). "Disruption of P bodies impairs mammalian RNA interference". Nat Cell Biol. 7 (12): 1267–74. doi:10.1038/ncb1334. PMID 16284622. S2CID 36630239.
- ^ Hammond S, Bernstein E, Beach D, Gregory Hannon (2000). "An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells". Nature. 404 (6775): 293–6. Bibcode:2000Natur.404..293H. doi:10.1038/35005107. PMID 10749213. S2CID 9091863. Greška u vankuverskom stilu: name u nazivu 4 (pomoć) Šablon:Closed access
- ^ Holmquist GP, Ashley T (2006). "Chromosome organization and chromatin modification: influence on genome function and evolution". Cytogenetic and Genome Research. 114 (2): 96–125. doi:10.1159/000093326. PMID 16825762. S2CID 29910065.
- ^ Verdel A, Jia S, Gerber S, Sugiyama T, Gygi S, Grewal SI, Moazed D (januar 2004). "RNAi-mediated targeting of heterochromatin by the RITS complex". Science. 303 (5658): 672–6. Bibcode:2004Sci...303..672V. doi:10.1126/science.1093686. PMC 3244756. PMID 14704433.
- ^ Irvine DV, Zaratiegui M, Tolia NH, Goto DB, Chitwood DH, Vaughn MW, Joshua-Tor L, Martienssen RA (august 2006). "Argonaute slicing is required for heterochromatic silencing and spreading". Science. 313 (5790): 1134–7. Bibcode:2006Sci...313.1134I. doi:10.1126/science.1128813. PMID 16931764. S2CID 42997104.
- ^ Volpe TA, Kidner C, Hall IM, Teng G, Grewal SI, Martienssen RA (septembar 2002). "Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi". Science. 297 (5588): 1833–7. Bibcode:2002Sci...297.1833V. doi:10.1126/science.1074973. PMID 12193640. S2CID 2613813.
- ^ Volpe T, Schramke V, Hamilton GL, White SA, Teng G, Martienssen RA, Allshire RC (2003). "RNA interference is required for normal centromere function in fission yeast". Chromosome Research. 11 (2): 137–46. doi:10.1023/A:1022815931524. PMID 12733640. S2CID 23813417.
- ^ Li LC, Okino ST, Zhao H, Pookot D, Place RF, Urakami S, Enokida H, Dahiya R (novembar 2006). "Small dsRNAs induce transcriptional activation in human cells". Proceedings of the National Academy of Sciences of the United States of America. 103 (46): 17337–42. Bibcode:2006PNAS..10317337L. doi:10.1073/pnas.0607015103. PMC 1859931. PMID 17085592.
- ^ Noma K, Sugiyama T, Cam H, Verdel A, Zofall M, Jia S, Moazed D, Grewal SI (novembar 2004). "RITS acts in cis to promote RNA interference-mediated transcriptional and post-transcriptional silencing". Nature Genetics. 36 (11): 1174–80. doi:10.1038/ng1452. PMID 15475954.
- ^ Sugiyama T, Cam H, Verdel A, Moazed D, Grewal SI (januar 2005). "RNA-dependent RNA polymerase is an essential component of a self-enforcing loop coupling heterochromatin assembly to siRNA production". Proceedings of the National Academy of Sciences of the United States of America. 102 (1): 152–7. Bibcode:2005PNAS..102..152S. doi:10.1073/pnas.0407641102. PMC 544066. PMID 15615848.
- ^ Wang F, Koyama N, Nishida H, Haraguchi T, Reith W, Tsukamoto T (juni 2006). "The assembly and maintenance of heterochromatin initiated by transgene repeats are independent of the RNA interference pathway in mammalian cells". Molecular and Cellular Biology. 26 (11): 4028–40. doi:10.1128/MCB.02189-05. PMC 1489094. PMID 16705157.
Vanjski linkovi
uredi- Overview of the RNAi process, from The Naked Scientists - Cambridge University's The Naked Scientists
- Animation of the RNAi process, from Nature
- NOVA scienceNOW explains RNAi – A 15-minute video of the Nova broadcast that aired on Public Broadcasting Service, 26 July 2005
- Silencing Genomes Arhivirano 10. 8. 2019. na Wayback Machine RNA interference (RNAi) experiments and bioinformatics in C. elegans for education. From the Dolan DNA Learning Center of Cold Spring Harbor Laboratory.
- RNAi screens in C. elegans in a 96-well liquid format and their application to the systematic identification of genetic interactions (a protocol)
- 2 American ‘Worm People’ Win Nobel for RNA Work, from NY Times
- Molecular Therapy web focus: "The development of RNAi as a therapeutic strategy", a collection of free articles about RNAi as a therapeutic strategy.
- GenomeRNAi: a database of phenotypes from RNA interference screening experiments in Drosophila melanogaster and Homo sapians
- RNAi tools Arhivirano 19. 6. 2018. na Wayback Machine Pre-designed and custom RNA Interference tools