Struktura proteina
Struktura proteina obuhvata svojstva rasporeda u prostoru molekula proteina koja proističu iz njihove aminokiselinske sekvence, fizičkih uvjeta u njihovoj okolini i prisustva jednostavnih ili složenih spojeva koji ih stabiliziraju i dovode do specifičnog presavijanja.[1]


Proteini su važna klasa bioloških makromolekula u svim biološkim organizmima, a čine ih elementi kao što su ugljik, vodik, dušik, kisik i sumpor. Svi proteini su polimerni lanci aminokiselina. Takvi polimeri, koji se nazivaju i peptidi, koji mogu sadržavati specifični broj i raspored kombinacije 20 različitih L-α-aminokiselina, opće formule RHC(NH2)COOH, čije se bočne funkcijske grupe (R) često nazivaju aminokiselinskim ostacima. Za lance ispod 40 ostataka, umesto naziva protein, upotreblava i se termin polipeptid. Za obavljanje njihove biološke funkcije, novonastali proteini se savijaju u jednu, ili više, specifičnih prostornih konformacija, u zavisnosti od broja nekovalentnih interakcija kao što su vodikova veza, ionske interakcije i Van der Valsove sile.[2] Da bi se spoznala funkcija proteina na molekulskoj razini, često je potrebno utvrditi njihovu trodimenzijsku strukturu. To je tema naučnog polja zvanog strukturna biologija, koja za utvrđivanje strukture proteina koristi tehnike kao što su rendgenska kristalografija ili NMR sprektroskopija.
Za obavljanje određenje biohemijske funkcije, neophodan je određeni broj ostataka, aoko 40-50 ostataka je pretpostavljena donja granica veličine djelotvornog domena. Veličina proteina kreće se od pomenute donje granice pa do nekoliko hiljada ostataka, u višefunkcijskim ili strukturnim proteinima. Ipak, sadašnja procjena za prosečnu dužinu proteina je oko 300 ostataka. Od proteinskih podjedinica, može se formirati veliki broj cjelina, npr. više hiljada molekula aktina, koji čine aktinska vlakna.
Razine i oblici strukture proteina
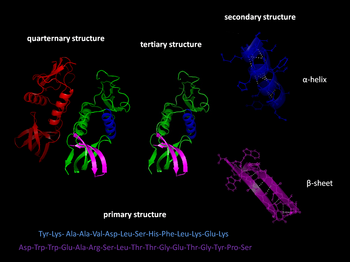
urediU biohemiji, razlikuju se četiri različita aspekta strukture proteina:
- Primarna struktura: aminokiselinska sekvenca peptidnog lanca (broj, vrsta i redosllijed aminokiselina u proteinu)
- Sekundarna struktura veoma je pravilne podstrukture – alfa-heliks i beta-list. Sekundarne strukture su definirane lokalno, što znači da mogu imati različite sekundarne motive u jednoj molekuli proteina.
- Tercijarna struktura je trodimenzijska struktura pojedinačnih molekula, kao prostorni raspored sekundarne strukture. Proces raskidanja tercijarne strukture može se pdvijati putem denaturacije.
- Kvaternerna struktura uključuje kompleks od nekoliko molekula proteina ili polipeptidnih lanaca, obično zvanih proteinska podjedinica, koji funkcionira kao dio većeg skupa ili proteinskog kompleksa.
U obavljanju bioloških funkcija, protein se može mijenjati preko nekoliko sličnih struktura. U kontekstu ovih funkcijskih preuređenja, tercijarne i kvaternerne strukture se obično nazivaju „konformacije“, a prijelazi između njih konformacijske promjene.
Primarnu strukturu odlikuje strukurna povezanost kovalentnim peptidnim vezama (hemijski gledano, to su amidne [[funkcionalna grupa|funkcijske grupe), koje nastaju tokom procesa translacije. Dva kraja lanca aminokiseline nazivaju se karboksilni kraj (C-kraj) i amino kraj (N-kraj), na osnovu prirode njihove slobodne grupe na svakom kraju.[2]
Različiti tipovi sekundarne strukture određeni su različitim šablonima vodikovih veza između glavnih lanaca peptidnih grupa. Ipak, ove veze općenito nisu u njima stabilne, pošto je vodikova veza voda–amid povoljnija nego amid–amid vodikova veza. Tačnije, sekundarna struktura je stabilna samo kad je lokalna koncentracija vode dovoljno velika, naprimjer, u potpuno savijenom stanju.
Slično, raspored globulastih proteina i tercijarne strukture je određen uglavnom strukturnim nespecifičnim interakcijama, kao što su sklonosti aminokiselina i hidrofobnih interakcija. Ipak, tercijarna struktura je određena samo onda kada su dijelovi proteinskog domena fiksirani pomoću specifičnih strukturnih interakcija, kao što su ionske interakcije, vodikove veze i čvrsto pakovanje na bočnim lancima. Tercijarna struktura vanćelijskih proteina može također biti stabilizirana pomoću disulfidnih veza, koje smanjuju entropiju opruženih oblika; disulfidni mostovi su izuzetno rijetki u citoplazmatskim proteinima, pošto je citoplazma općeredukcijsko okruženje.
Struktura aminokiselina
urediJedna α-aminokiselina sadrži dio, koji je zajednički za sve tipove aminokiselina, i bočni lanac (ostatak), koji je jedinstven za svaki od njih. α-C atom (Cα) je vezan za četiri različite grupe, jednu amino grupu, karboksilnu grupu, vodik i bočni lanac, specifičan za ovaj tip aminokiseline. Zato što je ugljikov atom vezan za četiri različite grupe, on je hiralan, a u biološkim proteinima nalazi se samo jedan od stereoizomera, predominantno L- oblik. Glicin pak, nije hiralan, pošto je njegov bočni lanac vodikov atom. Jednostavni mnemonik za pravilan L- oblik je CORN: kada se α-C atom posmatra tako da je H-atom ispred njega, ostaci se, u pravcu kretanja kazaljke na satu, mogu očitati kao CORN.
Bočni lanac utiče na hemijske osobine α-aminokiselina i može biti jedan od 20 različitih bočnih lanaca, kod aminokiselina koje tvore proteine (proteinogene aminokiseline).
Od 20 aminokiselina koje učestvuju u izgradnji proteina sve se uvrštavaju u neku od nekoliko grupa, na osnovu njihovih hemijskih osobina. Važni faktori su električni naboj, hidrofobnost/hidrofilnost, veličina i funkcijske grupe. Priroda interakcija različitih bočnih lacana sa vodenom okolinom igra glavnu ulogu u globularnoj proteinskoj strukturi. Hidrofobna strana lanca teži ka sidrenju u sredini proteina, dok je hidrofilna strana lanca izložena rastvaranju. Primjeri za hidrofobne ostatke su: leucin, izoleucin, fenilalanin i valin, a u manjoj mjeri tirozin, alanin i triptofan. Naelektrisanost bočnog lanca ima važnu ulogu u proteinskoj strukturi, pošto ionsko vezanje može stabilizirati proteinsku strukturu, dok neupareni naboj u sredini proteina može promijeniti strukturu. Naelektrisani ostaci su jako hidrofilni i obično se nalaze sa spoljne strane molekule proteina. Pozitivno naelektrisani bočni lanci su prisutni u lizinu i u argininu i, u nekim slučajevima, u histidinu. Negativna naelektrisanja imaju glutamin i aspartat. Ostatak aminokiselina ima manje hidrofilne bočne lance sa različitim funkcijskim grupama. Serin i treonin imaju hidroksilne grupe, a asparagin i glutamin – amidne grupe. Neke aminokiseline imaju specijalna svojstva, kao što su cistein, koji se, disulfidnom vezom, može kovalentno vezati, sa drugim cisteinima, prolin koji je cikličan, glicin, koji je mali i fleksibilniji od ostalih aminokiselina.
Peptidna veza
urediDvije aminokiseline mogu se kombinirati u reakcijama kondenzacije. Ponavljajući ovu reakciju, od ostataka, mogu nastati dugi lanci (amino kiseline u pepditnim vezama). Ova reakcija je katalizirana pomoću ribosoma, u procesu poznatom kao translacija. Peptidna veza je u suštini planarna, zahvaljujući delokalizaciji elektrona iz dvostruke veze. Za razliku od veoma krutog ugla peptidne veze, gdje je omega (veza između C1 i N) uvek blizu 180 stepeni, uglovi φ (veza između N i α-C) i Ψ (veza između α-C i C1) mogu imati određen domet mogućih vrijednosti. Ovi uglovi su stepeni slobode proteina, a kontrolišu trodimenzijsku strukturu proteina. Ograničeni su svojom geometrijom, da bi omogućili domete tipične za određene elemente sekundarne strukture, a predstavljeni su u Ramachandranovim dijagramom. Nekoliko važnih dužina veza prikazane su u sljedećoj tabeli.
| Peptidna veza | Prosječna dužina | Jednostruka veza | Prosječna dužina | Vodikova veza | Prosječno (±30) |
| Ca - C | 153 pm | C–C | 154 pm | O–H --- O-H | 280 pm |
| C–N | 133 pm | C–N | 148 pm | N–H --- O=C | 290 pm |
| N–Ca | 146 pm | C–O | 143 pm | O–H --- O=C | 280 pm |
Primarna struktura
urediNovonastala sekvenca različitih aminokiselina naziva se primarna struktura peptida ili proteina. Brojanje ostataka uvek počinje na završetku N-kraja (–NH2 grupa), koja je kraj gdje amino grupa koja je susjedna α-C atomu nije uključena u peptidnu vezu. Primarna struktura proteina je određena genom koji kodira protein. Specifična sekvenca nukleotida u DNK je prepisana na iRNK, koj „čitaju“ i „prevode“ ribosomi, u procesu translacije –završnog čina u biosintezi proteina. Niz aminokiselina je jedinstven za svaki protein i definira njegovu strukturu i funkciju. Određivanje primarne strukture proteina može se obaviti metodima kao što su Edmanova degradacija i masena spektroskopija. Često pak, može biti pročitana direktno sa gena koristeći genetički kod. Modifikacije nastale poslije transkripcije kao što su disulfidna formacija, fosforilacija i vezanje šećera obično se ostvaruju u dijelu primarne strukture i ne mogu biti pročitane sa gena.[3][4] Označavaju se kao posttranslacijske modifikacije
Sekundarna struktura
urediLinus Pauling sa saradnicima je 1951. predložio prve elemente sekundarne strukture, alfa-heliks i beta-konformaciju, pomoću modela peptida koje je napravio zahvaljujući poznatim informacijama o dužini veza i uglovima. I alfa-heliks i beta-konformacija predstavljaju način zasićenja svih donora i akceptora vodikovih veza u kičmi peptida.[5] Ovi elementi sekundarne strukture zavise samo od svojstava koja su zajednička za sve ostatke, objašnjavajući time zašto se često pojavljuju u većini proteina. Od tada su otkriveni i drugi elementi sekundarne strukture, kao što su različite petlje i drugi oblici heliksa. Dio kičme koji predstavlja pravu sekundarnu strukturu naziva se slučajna spirala. Svaki od ova dva sekundarna strukturna elementa ima pravilna geometrijska obilježja, što znači da su ograničeni specifičnim vrijednostima uglova φ i Ψ. Zato mogu biti pronađeni u specifičnoj oblasti Ramachandranove ravni.[6]
Tercijarna struktura
urediElementi sekundarne strukture obično se savijaju u kompaktan oblik, pomoću različitih spirala i uvijanja. Formacija tercijarne strukture obično se postiže pomoću usidravanja hidrofobnih ostataka, ali i druge interakcije, kao što su vodikove veze, ionske interakcije i disulfidne veze, mogu dovesti do stabilizacije tercijarne strukture. Tercijarna struktura zaokružuje sve nekovalentne interakcije, koje ne ostvaruje sekundarna struktura, definirajući sveukupno savijanje u proteinu i obično je nezamenjiva u funkciji proteina.
Kvaternerna struktura
urediKvaternerna struktura jostvaruje se interakcijom između nekoliko lanaca peptidnih veza. Pojedinačni lanci se nazivaju podjedinice. Pojedinačne podjedinice ne moraju biti kovalentno vezane , ali mogu imati disulfidne veze.[7] Ovu strukturu nemaju svi proteini, pošto mogu biti djelovati i kao monomeri. Kvaternerna struktura se stabilizira istim brojem interakcija kao i tercijarna. Kompleksi od dva ili više polimera nazivaju se multimeri. Ako sadrže dvije podjedinice, to su dimeri, a trimeri ako sadrže tri i tetrameri ako imaju četiri podjedinice. Multimeri od indentičnih podjedinica u nazivu imaju prefiks homo, dok oni koji su od različitih podjedinica imaju prefiks hetero.
Konformacija bočnog lanca
urediAtomi koji se nalaze duž bočnog lanca označavaju se slovima grčkog alfabeta, po njegovom redoslijedu: α, β, γ, δ i tako redom. Atom Cα (ili α-C) obično se smatra dijelom kičme. Diedarni uglovi oko međuatomskih veza nazivaju se π1, π2, π3 itd. Naprimjer, prvi i drugi ugljikov atom u bočnom lancu lizina su α i β, a diedarni uglovi oko α-β veze nazivaju se π1. Bočni lanac može se nalaziti u različitim konformacijama, označenim kao cis(–), trans i cis(+). Bočni lanci općenito nastoje da uđu u stepeničastu konformaciju oko π2, vođeni minimalizacijom preklapanja između elektronskih orbitala vodikovih atoma.
Motivi, domeni i savijanja proteinske strukture
urediMnogi proteini organizirani su u nekoliko jedinica. Strukturni domen je jedan od elemenata cjelokupne strukture proteina. To je je samostabilizirajuća struktura i često se savija nezavisno od ostatka proteinskog lanca. Mnogi domeni nisu jedinstveni proteinski proizvodi jednog gena ili porodice gena, već se mogu pojaviti u različitim proteinima. Domeni se često imenuju i izdvajaju jer imaju istaknutu ulogu u biološkoj funkciji proteina kojima pripadaju; naprimjer: kalcij-vezujući domen kalmudulina. Zato što su samostabilizirajući, domeni mogu biti zamenjeni postupcima genetičkog inženjerstva između dva proteina da bi se oformila himera. Pod motivom ovde se podrazumijeva mala specifična kombinacija sekundarnih elemenata strukture, koji se često nazivaju supersekundarne strukture. Savijanje se odnosi na globalni oblik savijanja. Strukturni motivi se obično sastoje od malog broja elemenata, na primer „uvijeni-heliks“ ima samo tri. Primjetno je da dok je prostorna sekvenca elemenata ista u svim slučajevima jednog motiva, ona može biti kodirana bilo kojom sekvencom osnovnovnog gena. Proteinski strukturni motivi često obuhvataju petlje različitih dužina i neočekivanih struktura, čiji je efekat da „popuste“ onoliko kolko je potrebno da bi se u prostoru spojila dva elementa koja nisu kodirana susjednim sekvencama DNK u genu. Takošer je uočeno da, iako dva gena istom sekvencom kodiraju sekundarne strukturalne elemente motiva, pored svega toga mogu specificirati nešto drugačiju sekvencu aminokiselina. U prilog ovome ide, ne samo kompliciran odnos između tercijalne i primarne strukture, već i to što veličina elemenata varira od jednog do drugog proteina. Uprkos tome što u eurokariotskim sistemima ima oko 100.000 različitih proteina, postoji mnogo manje različitih domena, strukturnih motiva i savijanja. To je djelimično posljedica evolucije, pošto geni ili njihovi dijelovi mogu biti duplirani ili „šetati“ unutar genoma. Ovo znači, naprimjer, da domen proteina može biti pomjeran od jednog do drugog proteina dajući proteinu novu funkciju. Zahvaljujući ovim putevima mehanizama, i sami mehanizmi teže da budu korišćeni u nekoliko različitih proteina.
Superdomen
urediSuperdomen sastoji se od dvije ili više nominalno nepovezanih strukturnih domena koji se nasljeđuju kao jedna cjelina i javljaju u različitim proteinima.[8] Primer su domen protein tirozin-fosfatazai C2 domenski par u PTEN-u, nekoliko tenzinskih proteina, auksilin i proteini kod biljaka i gljiva. Superdomen PTP-C2 očito je postojao prije divergencije gljiva, biljaka i životinja, pa je vjerovatno star oko 1,5 milijardi godina.
Dinamika proteina
urediProteini, međutim, nisu strogo statični objekti, već prije zauzimaju cjeline konformacijska stanja.
Prijelazi između ovih stanja obično se događaju na nanorazmjernoj skali s, i povezani su s funkcijski relevantnim pojavama, kao što je alosterna signalizacija[9] i enzimske katalize.[10] Dinamiku proteina i konformacijske promjene dopuštaju proteini koji funkcioniraju kao nanorazmjer [unutar bioloških mašina], unutar ćelija, često u obliku multiproteinskih kompleksA.[11] Primjeri uključuju motorne bjelančevine, kao što je miozin, koji je odgovoran za kontrakciju mišićA, kinezin, koji pomiče teret unutar ćelija dalje od [[Ćelijsko jedro]jedra]] duž mikrotubula i dineina, koji premješta teret unutar ćelija prema jedru i proizvodi aksonemano treperenje pokretnih treplji i bičeva. Učinak pokretnih treplji je nanomašina sastavljena od možda preko 600 proteina u molekulskim kompleksima, od kojih mnogi također funkcioniraju neovisno kao nanomašine. Fleksibilni linkeri omogućavaju mobilne proteinske domene koji su ih povezali, kako bi aktiviraliali svoje partnere za veanje i inducirali dugoročnu alostenost putem dinamika domena proteina.[12]
Savijanje proteina
urediProces kojim se stvaraju viši strukturni oblici naziva se proteinsko savijanje, a posljedica je primarne strukture. Jedinstveni polipeptid može imati više od jedne stabilne uvijene konformacije, koja može imati različitu biološku aktivnost, ali obično se samo jedna konformacija smatra aktivnom ili prirodnom konformacijom.[13]
Klasifikacija struktura
urediRazvijeno je nekoliko metoda za klasificiranje strukture proteina. Ova potraga teži i da klasifikuje podatke u Proteinskoj bazi podataka, po strukturnom redosledu. Postoji nekoliko baza podataka koje klasificiraju proteine koristeći različite metode. Najveće su SCOP, CATH i FSSP. Korišteni metod su čisto ručunarski, manipulativne i automatizirane ili čisto automatizirane. U toku su napori da bi se bolje razvrstali dosadašnji podaci. Klasifikacija je ista za većinu proteina koji su uvršteni i baze SCOP, CATH i FSSP, ali i dalje postoje neka neslaganja i nedosljednosti.[14][15]. Kada je strukturna sličnost velika, dva proteina su se možda odvojila od zajedničkog pretka[16], a zajednička struktura proteina smatra se dokazom homologijaijom. Tada se sličnost strukture može koristiti za grupiranje proteina u proteinske superporodice.[17]. Ako je zajednička struktura značajna, ali dijeljena frakcija je mala, taj fragment može biti posljedica dramatičnijeg evolucijskog događaja kao što je vodoravni prijenos gena, a spajanje proteina koji dijele ove fragmente u proteinske superfamilije više nije opravdano.[16] Za klasifikaciju proteina, može se koristiti i topologija proteina. Teorija čvorova i topologija kruga dva su okvira topologije razvijena za klasifikaciju proteinskih nabora na osnovu ukrštanja lanaca, odnosno unutarlančanih kontakata.
Određivanje strukture proteina
urediOko 90% struktura proteina koje su dostupne u proteinskim bazama podataka su određene pomoću kristalografije rendgenskim zračenjem. Ovaj metod omogućava mjerenje trodimenzijske gustine distribucije elektrona u proteinu, na osnovu čega se, sa određenom sigurnošću, izvode zaključci o 3D koordinatama svih atoma. Grubo procenjeno, oko 9% poznatih proteinskih struktura su dobijene pomoću tehnika nuklearne magnetne rezonanse, koje takođe mogu biti korištene da bi se odredila i sekundarna struktura. Potrebno je naglasiti da svojstvenosti sekundarne strukture mogu biti u cjelini određene i pomoću drugih biohemijskih tehnika kao što je cirkularni dihroizam. Sekundarna struktura također može biti predviđena sa visokom stopom preciznosti. Krio-elektronska mikroskopija je nedavno postala način određivanja proteinske strukture niske rezolucije. ali se predviđa da će u sljedećoj deceniji postati jedan od glavnih alata za određivanje u visokoj rezoluciji. Ova tehnika je još uvijek dragocjen izvor za istraživački rad sa veoma velikim proteinskim kompleksima, kao što su protein virusne opne i amiloidna vlakna.[18]
Računarsko predviđanje proteinske strukture
urediKreiranje proteinskih sekvenci je mnogo jednostavnije nego što je to za proteinske strukture. Ipak, proteinska struktura daje mnogo više podataka o unutrašnjoj funkciji proteina, nego njegova sekvenca. Zbog toga je predložen veći broj metoda za računarsko predviđanje proteinske strukture. Ab initio metode za predviđanje koriste samo sekvencu proteina. Metod računarske predikcije strukture proteina koristi postojeće proteinske strukture.[19]
Distribuirani kompjuterski projekt Roseta@home pokušava da predvidi proteinske strukture pomoću masivnog probanja na hiljadama kućnih računara.
Analiza proteinskih sekvenci
urediProteini se često smatraju relativno stabilnim strukturama koje imaju skup tercijarnih struktura i doživljavaju konformacijske promjene, kao rezultat modifikacije djelovanjem drugih proteina ili kao dio enzimske aktivnosti. Međutim, proteini imaju različit stupanj stabilnosti, a neke od manje stabilnih varijanti su suštinski poremećeni proteini. Ovi proteini postoje i funkcioniraju u relativno 'neuređenom' stanju bez stabilne tercijarne strukture. Kao rezultat toga, teško ih je opisati u standardnom modelu strukture proteina koji je dizajniran za proteine s fiksnom tercijarnom strukturom. Konformacijski ansambli osmišljeni su kao način da se pruži tačniji i 'dinamičniji' prikaz konformacijskog stanja suštinski poremećenih proteina. Funkcija konformacijskih ansambala pokušava predstaviti različite konformacije suštinski poremećenih proteina u datoteci ansambla (vrsta pronađena na /pedb.vib.be/ Baza podataka proteinskog ansambla).
Datoteke proteinskih ansambala su prikaz proteina za koji se može smatrati da imaju fleksibilnu strukturu. Stvaranje ovih datoteka zahtijeva utvrđivanje koje od različitih teorijski mogućih proteinskih konformacija zapravo postoje. Jedan od pristupa je primjena računskih algoritama za podatke o proteinima, kako bi se pokušalo odrediti najvjerovatniji skup konformacija za datoteku ansambla.
Postoji više metoda za pripremu podataka za [1] baze podataka proteinskog ansambla koje se dijele na dvije opća metoda: pristup bazenima podataka i molekulskoj dinamici (MD) (dijagramirani na slici). Pristup zasnovan na bazenu koristi aminokiselinsku sekvencu proteina, kako bi stvorio masivan skup slučajnih konformacija. Ovaj se bazen zatim podvrgava više računskoj obradi koja, na osnovu strukture, kreira skup teorijskih parametara za svaku konformaciju. Odabrani su konformacijski podskupovi iz ovog skupa čiji se prosječni teorijski parametri usko podudaraju s poznatim eksperimentalnim podacima za dati protein.[20]
Pristup molekulske dinamike uzima više slučajnih konformacija odjednom i sve ih podvrgava poređenju sa eksperimentalnim podacima. Ovdje eksperimentalni podaci služe kao ograničenja na postavljanje konformacija (npr. poznata udaljenost između atoma). Prihvataju se samo konformacije koje uspiju ostati u granicama postavljenim eksperimentalnim podacima. Ovaj pristup često primjenjuje velike količine eksperimentalnih podataka za konformacije, što je vrlo računarski zahtjevan zadatak..[20]
| Protein | Tip podataka | Protokol | PED ID | Reference |
|---|---|---|---|---|
| Sic1/Cdc4 | NMR i SAXS | Bazirani na bazenima | PED9AAA | [21] |
| p15 PAF | NMR i SAXS | Bazeni | PED6AAA[mrtav link] | [22] |
| MKK7 | NMR | Bazeni | PED5AAB Arhivirano 11. 9. 2016. na Wayback Machine | [23] |
| Beta-sinuklein | NMR | MD-baziran | PED1AAD Arhivirano 11. 9. 2016. na Wayback Machine | [24] |
| P27 KID | NMR | MD-baziran | PED2AAA Arhivirano 11. 9. 2016. na Wayback Machine | [25] |
(prema: image in "Computational approaches for inferring the functions of intrinsically disordered proteins"[20])
Također pogledajte
urediReference
uredi- ^ H. Stephen Stoker (1. 1. 2015). Organic and Biological Chemistry. Cengage Learning. str. 371. ISBN 978-1-305-68645-8.
- ^ a b Brocchieri L, Karlin S (10. 6. 2005). "Protein length in eukaryotic and prokaryotic proteomes". Nucleic Acids Research. 33 (10): 3390–3400. doi:10.1093/nar/gki615. PMC 1150220. PMID 15951512.
- ^ Sanger, F.; Tuppy, H. (1. 9. 1951). "The amino-acid sequence in the phenylalanyl chain of insulin. I. The identification of lower peptides from partial hydrolysates". The Biochemical Journal. 49 (4): 463–481. doi:10.1042/bj0490463. ISSN 0264-6021. PMC 1197535. PMID 14886310.
- ^ Sanger, F. (15. 5. 1959). "Chemistry of Insulin". Science (jezik: engleski). 129 (3359): 1340–1344. Bibcode:1959Sci...129.1340G. doi:10.1126/science.129.3359.1340. ISSN 0036-8075. PMID 13658959.
- ^ Pauling L, Corey RB, Branson HR (1951). "The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain". Proc Natl Acad Sci USA. 37 (4): 205–211. Bibcode:1951PNAS...37..205P. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
- ^ Chiang YS, Gelfand TI, Kister AE, Gelfand IM (2007). "New classification of supersecondary structures of sandwich-like proteins uncovers strict patterns of strand assemblage". Proteins. 68 (4): 915–921. doi:10.1002/prot.21473. PMID 17557333.
- ^ Moutevelis E, Woolfson DN (januar 2009). "A periodic table of coiled-coil protein structures". J. Mol. Biol. 385 (3): 726–32. doi:10.1016/j.jmb.2008.11.028. ISSN 0022-2836. PMID 19059267.
- ^ Haynie DT, Xue B (2015). "Superdomain in the protein structure hierarchy: the case of PTP-C2". Protein Science. 24 (5): 874–82. doi:10.1002/pro.2664. PMC 4420535. PMID 25694109.
- ^ Bu Z, Callaway DJ (2011). "Proteins MOVE! Protein dynamics and long-range allostery in cell signaling". Protein Structure and Diseases. Advances in Protein Chemistry and Structural Biology. 83. str. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 9780123812629. PMID 21570668.
- ^ Fraser JS, Clarkson MW, Degnan SC, Erion R, Kern D, Alber T (Dec 2009). "Hidden alternative structures of proline isomerase essential for catalysis". Nature. 462 (7273): 669–673. Bibcode:2009Natur.462..669F. doi:10.1038/nature08615. PMC 2805857. PMID 19956261.
- ^ Donald, Voet (2011). Biochemistry. Voet, Judith G. (4th izd.). Hoboken, NJ: John Wiley & Sons. ISBN 9780470570951. OCLC 690489261.
- ^ Satir, Peter; Søren T. Christensen (26. 3. 2008). "Structure and function of mammalian cilia". Histochemistry and Cell Biology. 129 (6): 687–93. doi:10.1007/s00418-008-0416-9. PMC 2386530. PMID 18365235. 1432-119X.
- ^ Govindarajan S, Recabarren R, Goldstein RA (17. 9. 1999). "Estimating the total number of protein folds". Proteins. 35 (4): 408–414. doi:10.1002/(SICI)1097-0134(19990601)35:4<408::AID-PROT4>3.0.CO;2-A. hdl:2027.42/34969. PMID 10382668. Arhivirano s originala, 5. 1. 2013.
- ^ Murzin, A. G.; Brenner, S.; Hubbard, T.; Chothia, C. (1995). "SCOP: A structural classification of proteins database for the investigation of sequences and structures" (PDF). Journal of Molecular Biology. 247 (4): 536–540. doi:10.1016/S0022-2836(05)80134-2. PMID 7723011. Arhivirano s originala (PDF), 26. 4. 2012.
- ^ Orengo, C; Michie, A. D.; Jones, S.; Jones, D. T.; Swindells, M. B.; Thornton, J. M. (1997). "CATH--a hierarchic classification of protein domain structures". Structure. 5 (8): 1093–1108. doi:10.1016/S0969-2126(97)00260-8. PMID 9309224.
- ^ a b Pascual-García, A.; Abia, D.; Ortiz, A.R.; Bastolla, U. (2009). "Cross-over between discrete and continuous protein structure space: insights into automatic classification and networks of protein structures". PLOS Computational Biology. 5 (3): e1000331. Bibcode:2009PLSCB...5E0331P. doi:10.1371/journal.pcbi.1000331. PMC 2654728. PMID 19325884.
- ^ Holm, L; Rosenström, P (juli 2010). "Dali server: conservation mapping in 3D". Nucleic Acids Research. 38 (Web Server issue): W545–9. doi:10.1093/nar/gkq366. PMC 2896194. PMID 20457744.
- ^ Laskowski, RA (2011). "Protein structure databases". Mol Biotechnol. 48 (2): 183–98. doi:10.1007/s12033-010-9372-4. PMID 21225378.
- ^ Zhang Y (2008). "Progress and challenges in protein structure prediction". Curr Opin Struct Biol. 18 (3): 342–348. doi:10.1016/j.sbi.2008.02.004. PMC 2680823. PMID 18436442.
- ^ a b c d Varadi, Mihaly; Vranken, Wim; Guharoy, Mainak; Tompa, Peter (1. 1. 2015). "Computational approaches for inferring the functions of intrinsically disordered proteins". Frontiers in Molecular Biosciences. 2: 45. doi:10.3389/fmolb.2015.00045. PMC 4525029. PMID 26301226.
- ^ Mittag, Tanja; Marsh, Joseph; Grishaev, Alexander; Orlicky, Stephen; Lin, Hong; Sicheri, Frank; Tyers, Mike; Forman-Kay, Julie D. (14. 3. 2010). "Structure/function implications in a dynamic complex of the intrinsically disordered Sic1 with the Cdc4 subunit of an SCF ubiquitin ligase". Structure. 18 (4): 494–506. doi:10.1016/j.str.2010.01.020. ISSN 1878-4186. PMC 2924144. PMID 20399186.
- ^ De Biasio, Alfredo; Ibáñez de Opakua, Alain; Cordeiro, Tiago N.; Villate, Maider; Merino, Nekane; Sibille, Nathalie; Lelli, Moreno; Diercks, Tammo; Bernadó, Pau (18. 2. 2014). "p15PAF is an intrinsically disordered protein with nonrandom structural preferences at sites of interaction with other proteins". Biophysical Journal. 106 (4): 865–874. Bibcode:2014BpJ...106..865D. doi:10.1016/j.bpj.2013.12.046. ISSN 1542-0086. PMC 3944474. PMID 24559989.
- ^ Kragelj, Jaka; Palencia, Andrés; Nanao, Max H.; Maurin, Damien; Bouvignies, Guillaume; Blackledge, Martin; Jensen, Malene Ringkjøbing (17. 3. 2015). "Structure and dynamics of the MKK7-JNK signaling complex". Proceedings of the National Academy of Sciences of the United States of America. 112 (11): 3409–3414. Bibcode:2015PNAS..112.3409K. doi:10.1073/pnas.1419528112. ISSN 1091-6490. PMC 4371970. PMID 25737554.
- ^ Allison, Jane R.; Rivers, Robert C.; Christodoulou, John C.; Vendruscolo, Michele; Dobson, Christopher M. (25. 11. 2014). "A relationship between the transient structure in the monomeric state and the aggregation propensities of α-synuclein and β-synuclein". Biochemistry. 53 (46): 7170–7183. doi:10.1021/bi5009326. ISSN 1520-4995. PMC 4245978. PMID 25389903.
- ^ Sivakolundu, Sivashankar G.; Bashford, Donald; Kriwacki, Richard W. (11. 11. 2005). "Disordered p27Kip1 exhibits intrinsic structure resembling the Cdk2/cyclin A-bound conformation". Journal of Molecular Biology. 353 (5): 1118–1128. doi:10.1016/j.jmb.2005.08.074. ISSN 0022-2836. PMID 16214166.