Sistem bakterijske sekrecije
Bakterijski sistemi lučenja su proteinski kompleksi na ćelijskim membranama bakterija za lučenje supstanci. Konkretno, oni su ćelijski uređaji koje koriste patogene bakterije za lučenje svojih faktora virulencije (uglavnom proteina) za invaziju na ćelije domaćina. Mogu se klasifikovati u različite tipove na osnovu njihove specifične strukture, sastava i aktivnosti. Općenito, proteini se mogu lučiti putem dva različita procesa. Jedan proces je mehanizam u jednom koraku, u kojem se proteini iz bakterijske citoplazme transportuju i isporučuju direktno kroz ćelijsku membranu u ćeliju domaćina. Drugi uključuje aktivnost u dva koraka u kojoj se proteini prvo transportuju iz unutrašnje ćelijske membrane, zatim se talože u perioplazmi i konačno kroz vanjsku ćelijsku membranu u ćeliju domaćina.[2]

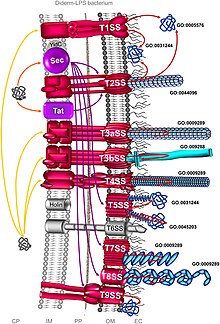
Ove velike razlike mogu se uočiti između Gram-negativne (diderm) i Gram-pozitivne bakterije (monoderm). Ali klasifikacija nikako nije jasna i potpuna. Postoji najmanje osam tipova specifičnih za Gram-negativne bakterije, četiri za Gram-pozitivne bakterije, dok su dvije zajedničke za oba tipa.[3] Osim toga, postoji značajna razlika između diderm bakterija sa lipopolisaharidom na vanjskoj membrani (diderm-LPS) i onih sa mikolnom kiselinom (diderm-mikolat).[1]
Putevi eksporta
urediEksportni put je odgovoran za prelazak unurtrašnje ćelijske membrane kod diderma i jedine ćelijske membrane kod monoderma.[1]
Sec sistem
urediOpća sekrecija (Sec) uključuje lučenje nesavijenih proteina koji prvo ostaju unutar ćelija. Kod Gram-negativnih bakterija, lučeni protein šalje se ili u unutrašnju membranu ili periplazmu. Ali kod Gram-pozitivnih bakterija, protein može ostati u ćeliji ili se uglavnom transportuje iz bakterije koristeći druge sisteme lučenja. Među Gram-negativnim bakterijama, Vibrio cholerae, Klebsiella pneumoniae i Yersinia enterocolitica koriste Sec sistem. Staphylococcus aureus i Listeria monocytogenes su Gram-pozitivne bakterije koje koriste Sec sistem.[4]
Sec sistem koristi dva različita puta za sekreciju: SecA i puteve čestica prepoznavanja signala (SRP). SecA je motorni protein ATPaza i ima mnogo srodnih proteina uključujući SecD, SecE, SecF, SegG, SecM i SecY. SRP je ribonukleoprotein (kompleks protein-RNK) koji prepoznaje i cilja specifične proteine na endoplazmatski retikulum kod eukariota i na ćelijsku membranu kod prokariota. Ova dva puta zahtijevaju različite molekulske šaperone i na kraju koriste kanal za transport proteina SecYEG za transport proteina kroz unutrašnju ćelijsku membranu.[5] U SecA putu, SecB djeluje kao šaperon, pomažući transportu proteina u periplazmu, nakon potpune sinteze peptidnih lanaca. Dok je u SRP putu, YidC pratilac i transportuje proteine do ćelijske membrane dok su još u toku sinteze peptida.[6]
SecA ili posttranslacijski put
urediProteini se sintetiziraju u ribosomima, procesom serijskog dodavanja aminokiselina, koji se naziva translacija. U SecA putu, faktor šaperonskog okidača (TF) se prvo vezuje za izloženu signalnu sekvencu N-terminalnog peptidnog lanca. Kako se produžavanje peptidnog lanca nastavlja, TF se zamjenjuje SecB. SecB specifično održava peptid u nesavijenom stanju i pomaže u vezivanju SecA. Kompleks se tada može vezati za SecYEG, čime se SecA aktivira vezivanjem sa ATP-om. Vođen energijom ATP-a, SecA gura protein kroz secYEG kanal. SecD/F kompleks također pomaže u povlačenju proteina s druge strane ćelijske membrane.[7]
SRP put
urediNa ovom putu, SRP se takmiči sa TF i vezuje se za signalnu sekvencu N-terminala. Proteini iz unutrašnje membrane zaustavljaju proces elongacije lanca. SRP se zatim vezuje za membranski receptor, FtsY. Kompleks peptidnog lanca-SRP-FtsY se zatim transportuje u SecY, gdje se nastavlja elongacija peptida.[6]
Tat sistem
urediSistem blizanačke translokacije arginina (Tat) sličan je Sec-u u procesu sekrecije proteina, međutim, šalje proteine samo u njihovom savijenom (tercijarnom) stanju. Koriste ga sve vrste bakterija, kao i arheje, te hloroplasti i biljne mitohondrije.[8] Kod bakterija, Tat sistem eksportuje proteine iz citoplazme kroz unutrašnju ćelijsku membranu; dok je u hloroplastima prisutan u tilakoidnoj membrani, gdje pomaže inos proteina iz strome.[9] Tat proteini su vrlo varijabilni u različitim bakterijama i klasificirani su u tri glavna tipa, a to su TatA, TatB i TatC. Naprimjer, dok postoje samo dva funkcionalna Tat proteina u Bacillus subtilis,[10] može ih biti preko stotinu u Streptomyces coelicolor.[11] Signalne peptide koji mogu prepoznati Tat-proteine karakterizira konsenzusni motiv Ser/Thr-Arg-Arg-X-Phe-Leu-Lys (gdje X može biti bilo koja polarna aminokiselina). To su dva uzastopna arginina od kojih potiče naziv argia blizansčka argininska translokacija. Zamjena bilo kojeg od arginina dovodi do usporavanja ili neuspjeha lučenja.[12]
Wss/Esx put
urediPut Wss/Esx (ESAT-6 sistem) naziva se ponekad i sistem sekrecije tipa VII (T7SS), iako je put eksporta.[1] Prisutan je u Gram-pozitivnim bakterijama (kao WSS) i kod Mycobacteria (kao Esx u svim diderm-mikolatima), kao što je M. tuberkuloza, M. bovis, Streptomyces coelicolor i S. aureus. Također se naziva T7b sistem u Bacillus subtilis i S. aureus. Sastoji se od dvije osnovne komponente: heksamerne ATPaze vezane za membranu koja je član porodice proteina FtsK/SpoIIIE,[13] i bilo koji od proteina povezanih s EsxA/EsxB, kao što su EsaA, EsaD, EsxB, EsxD, kao i Ess sistem (EssA, EssB i EsxC koji se nalaze u S. aureus).[14] EsxA i EsxB pripadaju nstporodicii WXG100 proteina koji formiraju dimerne spiralne ukosnice.
U S. aureus, T7SS luči veliki []toksin]] zvani EsaD, koji je član enzima nukleaza. EsaD se čini bezopasnim (detoksificiran) tokom svoje biosinteze uz pomoć svog analognog antitoksina EsaG. Kompleks EsaD-EsaG se tada vezuje za EsaE. EsaE dio se vezuje za EssC, koji je enzim ATPaza T7SS kompleksa. Tokom sekrecije, EsaG ostaje u citoplazmi, a zajedno se izlučuju samo EsaD i EsaE. Ali u nekim sojevima S. aureus, EsaD se ne proizvodi, već se umjesto toga formiraju dvije kopije proteina sličnih EsaG. Ovo bi moglo objasniti pojavu T7SS kod nepatogenih vrsta kao što je B. subtilis“ i „S. coelicolor.[15]
Sistemi sekrecije
urediSistemi sekrecije su odgovorni za prelazak vanjske ćelijske membrane ili obje membrane u diderma. Važećaa nomenklatura se odnosi samo na diderm-LPS, jer se ništa ne zna o tome šta diderm-mikolat bakterije koriste da pređu svoju vanjsku membranu.[1]
Tip I
urediHlyD koji se odmotava i kreće do vanjske ćelijske membrane. Terminalni signal prepoznaje TolC u unutrašnjoj membrani. HlyA se izlučuje iz vanjske membrane kroz proteinski kanal nalik tunelu.T1SS prenosi različite molekule uključujući ione, ugljikohidrate, lijekove, proteine. Izlučene molekule variraju u veličini od malog Escherichia coli peptidnog kolicina V, koji je 10 kDa, do ćelijskog adhezionog proteina LapA Pseudomonas fluorescens, koji ima 520 kDa.[16] Među najpoznatijim molekulama su RTX toksin i enzimi lipaze.
Tip II
urediSistem sekrecije tipa II (T2SS) zavisi od Sec ili Tat sistema za početno lučenje unutar bakterijske ćelije. Iz periplazme se proteini izlučuju iz sekretina vanjske membrane. Sekretini su multimerni (12-14 podjedinica) proteinski kompleksi koji formiraju pore. Sekretin je podržan od 10-15 ostalih proteina unutrašnje i vanjske membrane, koji čine kompletan aparat za lučenje.[17]
Tip III
urediSistem sekrecije tipa III (T3SS ili TTSS) je strukturno sličan i povezan sa baznim tijelom bakterijskih flagela. Vidi se u nekim od najvirulentnijih Gram-negativnih bakterija kao što su Salmonella, Shigella, Yersinia, Vibrio , koristi se za ubrizgavanje toksičnih proteina u eukariotske ćelije. Struktura T3SS se često opisuje kao injektizom ili aparat sličan igli/šprici. U Yersinia pestis, otkriveno je da T3SS može ubrizgati toksine direktno iz bakterijske citoplazme u citoplazmu ćelija svog domaćina.[18]
Tip IV
urediSistem sekrecije tipa IV (T4SS ili TFSS) povezan je sa sistemom bakterijske konjugacije, pomoću kojeg različite bakterije mogu razmjenjivati svoje DNK. Bakterije koje učestvuju mogu biti iste ili različite Gram-negativne bakterijske vrste. Može da transportuje pojedinačne proteine, kao i komplekse protein-protein i DNK-protein. Sekret se prenosi direktno iz ćelije primatelja kroz ćelijske membrane. Agrobacterium tumefaciens, iz kojeg je prvobitno otkriven, koristi ovaj sistem za slanje T-DNK dijela Ti-plazmida u biljne ćelije, u kojima se kao rezultat proizvodi krunska žuč (tumor). Helicobacter pylori ga koristi za isporuku CagA u epitelne ćelije želuca, da izazove rak želuca.[19] Bordetella pertussis, Uzročnik velikog kašlja, izlučuje svoj otrov pertusisa dijelom kroz T4SS. Legionella pneumophila koja uzrokuje legionelozu (legionarska bolest) ima T4SS koji se zove icm/dot, koji transportuju mnoge bakterijske proteine u svog eukariotskog domaćina.[20] Nedavno je pokazano da fitopatogen Xanthomonas citri koristi svoj T4SS za izlučivanje efektora koji su smrtonosni za druge bakterijske vrste, čime se ovaj sistem postavlja kao glavna determinanta fitnesa međuvrsne bakterijske konkurencije..[21][22] Prototipski tip IVA sekretaoni sistem je Virb kompleks Agrobacterium tumefaciens .[23]
Tip V
urediSistemi sekrecije tipa V (T5SS) razlikuju se od ostalih sistema sekrecije po tome što se sami luče i uključuju samo vanjsku ćelijsku membranu. Da bi izlučeni protein prošao kroz unutrašnju ćelijsku membranu, T5SS zavisi od Sec sistema. Imaju domen β-barela, koji se ubacuje u vanjsku ćelijsku membranu i formira kanal koji može transportirati lučeni protein zajedno s njim. Zbog ovakve aktivnosti nazivaju se i autotransporterski sistemi.[24] Kad su izlučeni proteini izloženi vani, autotransporteri su odsječeni (otcijepljeni), oslobađajući protein iz domene β-barela. Primjer autotransportera je trimerni autotransporter adezina (TAA).[25]
TIP VI
urediTip VI sistema sekrecije (T6SS) je otkrio tim Johna Mekalanosa na Harvard Medicinskoj školi 2006. godine kod Vibrio cholerae i Pseudomonas aeruginosa.[26][27] Prepoznati su kada su mutacije u genima Vibrio cholerae Hcp i VrgG uzrokovale smanjenu virulentnost i patogenost.[28][29] In addition to their classic role as the pathogenicity factor, T6SS are also involved in defense against simple eukaryotic predators and in inter-bacteria interactions.[30][31] Gen za T6SS formira klaster gena koji se sastoji od više od 15 gena. Geni Hcp i VgrG su najuniverzalniji geni. Strukturna sličnost T6SS sa repom faga T4 sugeriše da je proces infekcije sličan onom kod faga.[32]
Tip VII
urediT7SS diderm-LPS bakterija je Chaperone-Usherov put (CUP).[1]
Tip VIII
urediT8SS diderm-LPS bakterija je put vanćelijske nukleacije-precipitacije (ENP).[1]
Tip IX
urediSistemi sekrecije tipa IX (T9SS) nalaze se redovno u lozi bakterija Fibrobacteres-Chlorobi-Bacteroidetes, gdje vrste članova uključuju vanjsku membranu. Sistem je različito uključen u jednu vrstu pokretljivosti klizanja, u pravilno ciljanje određenih faktora virulencije na površinu ćelije i degradaciju kompleksa biopolimera.[33] T9SS je također poznat kao lučenje Por (akumulacija porfirina na površini ćelije),[1] prema oralnom patogenu Porphyromonas gingivalis. Opisano je najmanje šesnaest strukturnih komponenti sistema, uključujući PorU, transpeptidaza sortiranja proteina koja uklanja signal za sortiranje C-terminalnog proteina iz teretnih proteina i posreduje u njihovom vezivanju za lipopolisaharide.
Reference
uredi- ^ a b c d e f g h i Chagnot, C; Zorgani, MA; Astruc, T; Desvaux, M (14 October 2013). "Proteinaceous determinants of surface colonization in bacteria: bacterial adhesion and biofilm formation from a protein secretion perspective". Frontiers in Microbiology. 4: 303. doi:10.3389/fmicb.2013.00303. PMC 3796261. PMID 24133488.
- ^ Bocian-Ostrzycka KM, Grzeszczuk MJ, Banaś AM, Jagusztyn-Krynicka EK (May 2017). "Bacterial thiol oxidoreductases - from basic research to new antibacterial strategies". Applied Microbiology and Biotechnology. 101 (10): 3977–3989. doi:10.1007/s00253-017-8291-8. PMC 5403849. PMID 28409380.
- ^ Green ER, Mecsas J (February 2016). Kudva IT (ured.). "Bacterial Secretion Systems: An Overview". Microbiology Spectrum (5 izd.). American Society for Microbiology Press. 4 (1): 215–239. doi:10.1128/microbiolspec.VMBF-0012-2015. ISBN 9781555819286. PMC 4804464. PMID 26999395.
- ^ Bensing BA, Seepersaud R, Yen YT, Sullam PM (August 2014). "Selective transport by SecA2: an expanding family of customized motor proteins". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1843 (8): 1674–86. doi:10.1016/j.bbamcr.2013.10.019. PMC 4007388. PMID 24184206.
- ^ Crane JM, Randall LL (November 2017). "Escherichia coli". EcoSal Plus. 7 (2): ESP–0002–2017. doi:10.1128/ecosalplus.ESP-0002-2017. PMC 5807066. PMID 29165233.
- ^ a b Zhu L, Kaback HR, Dalbey RE (September 2013). "YidC protein, a molecular chaperone for LacY protein folding via the SecYEG protein machinery". The Journal of Biological Chemistry. 288 (39): 28180–94. doi:10.1074/jbc.M113.491613. PMC 3784728. PMID 23928306.
- ^ Lycklama A, Nijeholt JA, Driessen AJ (April 2012). "The bacterial Sec-translocase: structure and mechanism". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 367 (1592): 1016–28. doi:10.1098/rstb.2011.0201. PMC 3297432. PMID 22411975.
- ^ Yen MR, Tseng YH, Nguyen EH, Wu LF, Saier MH (June 2002). "Sequence and phylogenetic analyses of the twin-arginine targeting (Tat) protein export system". Archives of Microbiology. 177 (6): 441–50. doi:10.1007/s00203-002-0408-4. PMID 12029389. S2CID 25129008.
- ^ Lee PA, Tullman-Ercek D, Georgiou G (2006). "The bacterial twin-arginine translocation pathway". Annual Review of Microbiology. 60: 373–95. doi:10.1146/annurev.micro.60.080805.142212. PMC 2654714. PMID 16756481.
- ^ Jongbloed JD, Grieger U, Antelmann H, Hecker M, Nijland R, Bron S, van Dijl JM (December 2004). "Two minimal Tat translocases in Bacillus". Molecular Microbiology. 54 (5): 1319–25. doi:10.1111/j.1365-2958.2004.04341.x. PMID 15554971.
- ^ Li H, Jacques PE, Ghinet MG, Brzezinski R, Morosoli R (July 2005). "Determining the functionality of putative Tat-dependent signal peptides in Streptomyces coelicolor A3(2) by using two different reporter proteins". Microbiology. 151 (Pt 7): 2189–98. doi:10.1099/mic.0.27893-0. PMID 16000709.
- ^ Stanley NR, Palmer T, Berks BC (April 2000). "The twin arginine consensus motif of Tat signal peptides is involved in Sec-independent protein targeting in Escherichia coli". The Journal of Biological Chemistry. 275 (16): 11591–6. doi:10.1074/jbc.275.16.11591. PMID 10766774.
- ^ Aly KA, Anderson M, Ohr RJ, Missiakas D (December 2017). "Isolation of a Membrane Protein Complex for Type VII Secretion in Staphylococcus aureus". Journal of Bacteriology. 199 (23): e00482–17. doi:10.1128/JB.00482-17. PMC 5686593. PMID 28874412.
- ^ Kneuper H, Cao ZP, Twomey KB, Zoltner M, Jäger F, Cargill JS, et al. (September 2014). "Heterogeneity in ess transcriptional organization and variable contribution of the Ess/Type VII protein secretion system to virulence across closely related Staphylocccus aureus strains". Molecular Microbiology. 93 (5): 928–43. doi:10.1111/mmi.12707. PMC 4285178. PMID 25040609.
- ^ Cao Z, Casabona MG, Kneuper H, Chalmers JD, Palmer T (October 2016). "The type VII secretion system of Staphylococcus aureus secretes a nuclease toxin that targets competitor bacteria". Nature Microbiology. 2: 16183. doi:10.1038/nmicrobiol.2016.183. PMC 5325307. PMID 27723728.
- ^ Boyd CD, Smith TJ, El-Kirat-Chatel S, Newell PD, Dufrêne YF, O'Toole GA (August 2014). "Structural features of the Pseudomonas fluorescens biofilm adhesin LapA required for LapG-dependent cleavage, biofilm formation, and cell surface localization". Journal of Bacteriology. 196 (15): 2775–88. doi:10.1128/JB.01629-14. PMC 4135675. PMID 24837291.
- ^ Korotkov KV, Sandkvist M, Hol WG (April 2012). "The type II secretion system: biogenesis, molecular architecture and mechanism". Nature Reviews. Microbiology. 10 (5): 336–51. doi:10.1038/nrmicro2762. PMC 3705712. PMID 22466878.
- ^ Büttner D (June 2012). "Protein export according to schedule: architecture, assembly, and regulation of type III secretion systems from plant- and animal-pathogenic bacteria". Microbiology and Molecular Biology Reviews. 76 (2): 262–310. doi:10.1128/MMBR.05017-11. PMC 3372255. PMID 22688814.
- ^ Hatakeyama M, Higashi H (December 2005). "Helicobacter pylori CagA: a new paradigm for bacterial carcinogenesis". Cancer Science. 96 (12): 835–43. doi:10.1111/j.1349-7006.2005.00130.x. PMID 16367902. S2CID 5721063.
- ^ Cascales E, Christie PJ (November 2003). "The versatile bacterial type IV secretion systems". Nature Reviews. Microbiology. 1 (2): 137–49. doi:10.1038/nrmicro753. PMC 3873781. PMID 15035043.
- ^ Souza DP, Oka GU, Alvarez-Martinez CE, Bisson-Filho AW, Dunger G, Hobeika L, et al. (March 2015). "Bacterial killing via a type IV secretion system". Nature Communications. 6: 6453. Bibcode:2015NatCo...6.6453S. doi:10.1038/ncomms7453. PMID 25743609.
- ^ Sgro GG, Costa TR, Cenens W, Souza DP, Cassago A, Coutinho de Oliveira L, et al. (December 2018). "Cryo-EM structure of the bacteria-killing type IV secretion system core complex from Xanthomonas citri". Nature Microbiology. 3 (12): 1429–1440. doi:10.1038/s41564-018-0262-z. PMC 6264810. PMID 30349081.
- ^ Christie PJ, Atmakuri K, Krishnamoorthy V, Jakubowski S, Cascales E (2005). "Biogenesis, architecture, and function of bacterial type IV secretion systems". Annual Review of Microbiology. 59: 451–85. doi:10.1146/annurev.micro.58.030603.123630. PMC 3872966. PMID 16153176.
- ^ Thanassi DG, Stathopoulos C, Karkal A, Li H (2005). "Protein secretion in the absence of ATP: the autotransporter, two-partner secretion and chaperone/usher pathways of gram-negative bacteria (review)". Molecular Membrane Biology. 22 (1–2): 63–72. doi:10.1080/09687860500063290. PMID 16092525. S2CID 2708575.
- ^ Gerlach RG, Hensel M (October 2007). "Protein secretion systems and adhesins: the molecular armory of Gram-negative pathogens". International Journal of Medical Microbiology. 297 (6): 401–15. doi:10.1016/j.ijmm.2007.03.017. PMID 17482513.
- ^ Pukatzki S, Ma AT, Sturtevant D, Krastins B, Sarracino D, Nelson WC, et al. (January 2006). "Identification of a conserved bacterial protein secretion system in Vibrio cholerae using the Dictyostelium host model system". Proceedings of the National Academy of Sciences of the United States of America. 103 (5): 1528–33. Bibcode:2006PNAS..103.1528P. doi:10.1073/pnas.0510322103. JSTOR 30048406. PMC 1345711. PMID 16432199.
- ^ Mougous JD, Cuff ME, Raunser S, Shen A, Zhou M, Gifford CA, et al. (June 2006). "A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus". Science. 312 (5779): 1526–30. Bibcode:2006Sci...312.1526M. doi:10.1126/science.1128393. PMC 2800167. PMID 16763151.
- ^ Bingle LE, Bailey CM, Pallen MJ (February 2008). "Type VI secretion: a beginner's guide" (PDF). Current Opinion in Microbiology. 11 (1): 3–8. doi:10.1016/j.mib.2008.01.006. PMID 18289922.
- ^ Cascales E (August 2008). "The type VI secretion toolkit". EMBO Reports. 9 (8): 735–41. doi:10.1038/embor.2008.131. PMC 2515208. PMID 18617888.
- ^ Schwarz S, Hood RD, Mougous JD (December 2010). "What is type VI secretion doing in all those bugs?". Trends in Microbiology. 18 (12): 531–7. doi:10.1016/j.tim.2010.09.001. PMC 2991376. PMID 20961764.
- ^ Coulthurst SJ (2013). "The Type VI secretion system - a widespread and versatile cell targeting system". Research in Microbiology. 164 (6): 640–54. doi:10.1016/j.resmic.2013.03.017. PMID 23542428.
- ^ Silverman JM, Brunet YR, Cascales E, Mougous JD (2012). "Structure and regulation of the type VI secretion system". Annual Review of Microbiology. 66: 453–72. doi:10.1146/annurev-micro-121809-151619. PMC 3595004. PMID 22746332.
- ^ Veith PD, Glew MD, Gorasia DG, Reynolds EC (October 2017). "Type IX secretion: the generation of bacterial cell surface coatings involved in virulence, gliding motility and the degradation of complex biopolymers". Molecular Microbiology. 106 (1): 35–53. doi:10.1111/mmi.13752. hdl:11343/208056. PMID 28714554. S2CID 19387266.