Ankirinsko ponavljanje
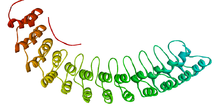
Ankirinsko ponavljanje je motiv od 33 aminokiselinska ostatka u proteinima koji se sastoji od dva alfa-heliksa odvojenih petljom. Prvi put otkriveno je u signaliziranju proteina u kvašćevom Cdc10 i Notch-u u roduDrosophila. Domeni koji se sastoje od ankirinskih tandemskih ponavljanja i posreduju u interakcijama protein-protein, a među najčešćim su strukturnim motivima u poznatim proteinima. Pojavljuju se u proteinima Bacteria, Archaeal i Eukaryota, ali su daleko najčešći kod eukariota., Iako ih nema u većini virusa, ankirinski ponavljajući proteini uobičajeni su među poksvirusima. Većina proteina koji sadrže motiv ima četiri do šest ponavljanja, iako ih njegov imenjak ankirin sadrži 24, a najveći poznati broj ponavljanja je 34, što je predviđeno u proteinu izraženom kod Giardia lamblia.[2]
| Domen ankirinskog ponavlja | |
|---|---|
 | |
| Identifikatori | |
| Simbol | Ank |
| PDB | [http://www.rcsb.org/pdb/cgi/explore.cgi?pdbId=1a5e 1a5e, 1ap7, 1awc, 1bd8, 1bi7, 1bi8, 1blx, 1bu9, 1d9s, 1dc2, 1dcq, 1g3n, 1ihb, 1ikn, 1ixv, 1k1a, 1k1b, 1k3z, 1mx2, 1mx4, 1mx6, 1myo, 1n11, 1nfi, 1ot8, 1oy3, 1qym, 1s70, 1svc, 1sw6, 1tr4, 1uoh, 1wdy, 1wfu, 1wg0, 1ycs, 1yyh, 1zlm, 2a5e, 2f8x, 2f8y, 2he0, 2myo 1a5e, 1ap7, 1awc, 1bd8, 1bi7, 1bi8, 1blx, 1bu9, 1d9s, 1dc2, 1dcq, 1g3n, 1ihb, 1ikn, 1ixv, 1k1a, 1k1b, 1k3z, 1mx2, 1mx4, 1mx6, 1myo, 1n11, 1nfi, 1ot8, 1oy3, 1qym, 1s70, 1svc, 1sw6, 1tr4, 1uoh, 1wdy, 1wfu, 1wg0, 1ycs, 1yyh, 1zlm, 2a5e, 2f8x, 2f8y, 2he0, 2myo] |
Ankirin se tipično ponavlja zajedno sa savijanjem da formira jednu, linearnu solenoidnu strukturu zvanu ankirinski ponovljeni domeni. Ovi domeni su jedna od najčešćih platformi za interakcije protein-protein u prirodi. Javljaju se u velikom broju funkcionalno raznolikih proteina, uglavnom iz eukariotskih. Nekoliko poznatih primjera prokariotakih i virusnih može biti posljedica horizontalnog transfera gena.[3] Ponavljanje je pronađeno u proteinima različitih funkcija kao što su inicijatori transkripcije, regulatori ćelijskog ciklusa, citoskeletni, ionski transporteri i transduktori signala. Čini se da je ankirinsko savijanje definirano svojom strukturom, a ne funkcijom, budući da ne postoji specifična sekvenca ili struktura koja bi bila univerzalno prepoznata po njemu.
Uzimajući u obzir atomske strukture pojedinačnih ankirinskih ponavljanja, petlja je često tipa 1 beta izbočena petlja, dok oba alfa-heliksa na svom N-terminalu obično imaju Schellmanovu petlju.
Uloga u savijanju proteina
uredimotiv sekvence ponavljanja ankirina proučavan je korištenjem poravnavanja višestrukih sekvenci da bi se odredila konzerviranost aminokiselinskih ostaka kritičnih za savijanje i stabilnost. Ostaci na širokoj bočnoj površini ankirinskih ponavljajućih struktura su promjenjivi, često hidrofobni i uključeni uglavnom u posredovanje interakcija protein-protein. Vještački dizajn proteina zasnovan na konsenzusnoj sekvenci izvedenoj iz poravnanja sekvenci sintetiziran je i utvrđeno je da se savija stabilno, predstavljajući prvi dizajnirani protein s višestrukim ponavljanjima.[4] Opsežnije strategije dizajna koristile su kombinatorne sekvence za "evoluciju" ankirinskih ponavljanja koja prepoznaju određene proteinske mete, tehnika koja je predstavljena kao alternativa dizajnu antitijela za aplikacije koje zahtijevaju vezivanje visokog afiniteta.[5] Studija zasnovana na strukturi koja uključuje niz ankirinskih proteina poznatih struktura, pokazuje da su ti proteini zasnovani na konsenzusu vrlo stabilni jer maksimiziraju energetski jaz između struktura koje se sklapaju i rasklapaju, kodirajući gusto povezanu mrežu povoljnih interakcija između motiva konzertviranih sekvenci, poput motiva TPLX.[6] Ista studija pokazuje da su insercije u kanonski okvir ankirinskih ponavljanja obogaćena konfliktnim interakcijama, koje su vezane za funkciju. Isto se odnosi i na interakcije oko žarišta za deleciju. Ovo može biti povezano sa složenim prijelazima savijanja/odmotavanja koji su važni za prepoznavanje i interakciju partnera.
Ankirinski ponavljajući proteini predstavljaju neobičan problem u proučavanju savijanja proteina, koji se u velikoj mjeri fokusirao na globulaste proteine koji formiraju dobro definiranu tercijarnu strukturu stabiliziranu dalekometnim, nelokalnim kontaktima ostatak-ostatak. Ankirinska ponavljanja, nasuprot tome, sadrže vrlo malo takvih kontakata (to jest, imaju nizak redoslijed kontakta). Većina studija otkrila je da se ankirin ponavlja savijanjem u mehanizmu sklapanje u dva stanja, što ukazuje na visok stepen kooperativnosti savijanja uprkos lokalnim kontaktima između ostataka i evidentnu potrebu za uspješnim savijanjem s različitim brojem ponavljanja. Neki dokazi, zasnovani na sintezi skraćenih verzija prirodnih ponovljenih proteina,[7] i o ispitivanju phi vrijednosti,[8] sugerira da C-terminal formira mjesto preklapanja nukleacije.
Klinički značaj
urediProteini koji ankirinskog ponavljanja povezani su sa brojnim ljudskim bolestima. Ovi proteini uključuju inhibitor ćelijskog ciklusa p16, koji je povezan sa rakom, i Notch protein (ključna komponenta ćelijskih signalnih puteva) koji može uzrokovati neurološke poremećaj CADASIL kada je domen ponavljanja poremećen mutacijama.[2]
Specijalizirana porodica ankirinskih proteina poznatih kao mišićni ankirin ponavljajući proteini (MARP) uključeni su u popravku i regeneraciju mišićnog tkiva nakon oštećenja uslijed ozljede i stresa.[9]
Prirodna varijacija između glutamina i lizina na poziciji 703 u 11. ankirinskom ponavljanju ANKK1, poznata kao TaqI A1 alel,[10] zaslužna je za podršku ovisnosti kao što su gojaznost, alkoholizam, ovisnost o nikotinu i spolnoj aktivnost, dok obeshrabruje maloljetničku delinkvenciju i anksioznost neuroticizma.[11]
Ljudski proteini sa ankirinskim ponavljanjem
urediABTB1; ABTB2; ACBD6; ACTBL1; ANK1; ANK2; ANK3; ANKAR; ANKDD1A; ANKEF1; ANKFY1; ANKHD1; ANKIB1; ANKK1; ANKMY1; ANKMY2; ANKRA2; ANKRD1; ANKRD10; ANKRD11; ANKRD12; ANKRD13; ANKRD13A; ANKRD13B; ANKRD13C; ANKRD13D; ANKRD15; ANKRD16; ANKRD17; ANKRD18A; ANKRD18B; ANKRD19; ANKRD2; ANKRD20A1; ANKRD20A2; ANKRD20A3; ANKRD20A4; ANKRD21; ANKRD22; ANKRD23; ANKRD24; ANKRD25; ANKRD26; ANKRD27; ANKRD28; ANKRD30A; ANKRD30B; ANKRD30BL; ANKRD32; ANKRD33; ANKRD35; ANKRD36; ANKRD36B; ANKRD37; ANKRD38; ANKRD39; ANKRD40; ANKRD41; ANKRD42; ANKRD43; ANKRD44; ANKRD45; ANKRD46; ANKRD47; ANKRD49; ANKRD50; ANKRD52; ANKRD53; ANKRD54; ANKRD55; ANKRD56; ANKRD57; ANKRD58; ANKRD60; ANKRD6; ANKRD7; ANKRD9; ANKS1A; ANKS3; ANKS4B; ANKS6; ANKZF1; ASB1; ASB10; ASB11; ASB12; ASB13; ASB14; ASB15; ASB16; ASB2; ASB3; ASB4; ASB5; ASB6; ASB7; ASB8; ASB9; ASZ1; BARD1; BAT4; BAT8; BCL3; BCOR; BCORL1; BTBD11; CAMTA1; CAMTA2; CASKIN1; CASKIN2; CCM1; CDKN2A; CDKN2B; CDKN2C; CDKN2D; CENTB1; CENTB2; CENTB5; CENTG1; CENTG2; CENTG3; CLIP3; CLIP4; CLPB; CTGLF1; CTGLF2; CTGLF3; CTGLF4; CTGLF5; CTTNBP2; DAPK1; DDEF1; DDEF2; DDEFL1; DGKI; DGKZ; DP58; DYSFIP1; DZANK; EHMT1; EHMT2; ESPN; FANK1; FEM1A; FEM1B; GABPB2; GIT1; GIT2; GLS; GLS2; HACE1; HECTD1; IBTK; ILK; INVS; KIDINS220; KRIT1; LRRK1; MAIL; MIB1; MIB2; MPHOSPH8; MTPN; MYO16; NFKB1; NFKB2; NFKBIA; NFKBIB; NFKBIE; NFKBIL1; NFKBIL2; NOTCH1; NOTCH2; NOTCH3; NOTCH4; NRARP; NUDT12; OSBPL1A; OSTF1; PLA2G6; POTE14; POTE15; POTE8; PPP1R12A; PPP1R12B; PPP1R12C; PPP1R13B; PPP1R13L; PPP1R16A; PPP1R16B; PSMD10; RAI14; RFXANK; RIPK4; RNASEL; SHANK1; SHANK2; SHANK3; SNCAIP; TA-NFKBH; TEX14; TNKS; TNKS2; TNNI3K; TP53BP2; TRP7; TRPA1; TRPC3; TRPC4; TRPC5; TRPC6; TRPC7; TRPV1; TRPV2; TRPV3; TRPV4; TRPV5; TRPV6; UACA; USH1G; ZDHHC13; ZDHHC17;
Također pogledajte
urediReference
uredi- ^ PDB 1N11; Michaely P, Tomchick DR, Machius M, Anderson RG (decembar 2002). "Crystal structure of a 12 ANK repeat stack from human ANK1". EMBO J. 21 (23): 6387–96. doi:10.1093/emboj/cdf651. PMC 136955. PMID 12456646.
- ^ a b Mosavi L, Cammett T, Desrosiers D, Peng Z (2004). "The ankyrin repeat as molecular architecture for protein recognition". Protein Sci. 13 (6): 1435–48. doi:10.1110/ps.03554604. PMC 2279977. PMID 15152081. Arhivirano s originala, 7. 9. 2004.
- ^ Bork P (decembar 1993). "Hundreds of ankyrin-like repeats in functionally diverse proteins: mobile modules that cross phyla horizontally?". Proteins. 17 (4): 363–74. doi:10.1002/prot.340170405. PMID 8108379. S2CID 35224626.
- ^ Mosavi LK, Minor DL, Peng ZY (Dec 2002). "Consensus-derived structural determinants of the ankyrin repeat motif". Proc Natl Acad Sci USA. 99 (25): 16029–34. Bibcode:2002PNAS...9916029M. doi:10.1073/pnas.252537899. PMC 138559. PMID 12461176.
- ^ Binz HK, Amstutz P, Kohl A, et al. (maj 2004). "High-affinity binders selected from designed ankyrin repeat protein libraries". Nat. Biotechnol. 22 (5): 575–82. doi:10.1038/nbt962. PMID 15097997. S2CID 1191035.
- ^ Parra RG, Espada R, Verstraete N, Ferreiro DU, et al. (Dec 2015). "Structural and Energetic Characterization of the Ankyrin Repeat Protein Family". PLOS Comput. Biol. 12 (11): 575–82. Bibcode:2015PLSCB..11E4659P. doi:10.1371/journal.pcbi.1004659. PMC 4687027. PMID 26691182.
- ^ Zhang B, Peng Z (Jun 2000). "A minimum folding unit in the ankyrin repeat protein p16(INK4)". J Mol Biol. 299 (4): 1121–32. doi:10.1006/jmbi.2000.3803. PMID 10843863.
- ^ Tang KS, Fersht AR, Itzhaki LS (Jan 2003). "Sequential unfolding of ankyrin repeats in tumor suppressor p16". Structure. 11 (1): 67–73. doi:10.1016/S0969-2126(02)00929-2. PMID 12517341.
- ^ Miller MK, Bang ML, Witt CC, et al. (Nov 2003). "The muscle ankyrin repeat proteins: CARP, ankrd2/Arpp and DARP as a family of titin filament-based stress response molecules". J Mol Biol. 333 (5): 951–64. doi:10.1016/j.jmb.2003.09.012. PMID 14583192.
- ^ Neville MJ, Johnstone EC, Walton RT (Jun 2004). "Identification and characterization of ANKK1: a novel kinase gene closely linked to DRD2 on chromosome band 11q23.1". Hum. Mutat. 23 (6): 540–5. doi:10.1002/humu.20039. PMID 15146457. S2CID 22242611.
- ^ "NCBI Gene summary for DRD2". (interim reference)
Vanjsli linkovi
urediEukaryotic Linear Motif resource motif class LIG_TNKBM_1
- Ankyrin repeat na US National Library of Medicine Medical Subject Headings (MeSH)