Komplementarnost (molekularna biologija)
U molekulskoj biologiji, pojam komplementarnost podrazumijeva takve odnose između dvije strukture koje se poklapaju po obrascu brave i ključa. U prirodi, komplementarnost je osnovni princip replikacije DNK i transkripcije, jer je to svojstvo koje dijele idvije sekvence sekvence DNK ili RNK, tako da kada su antiparalelno međusobno poravnate, nukleotidne baze na svakom položaju u sekvencama će biti komplementarne, slično kao da se gleda u ogledalo i vidi naličje stvari. Ovo komplementarno uparivanje baza omogućava ćelijama da kopira informacije iz jedne generacije u drugu, pa čak i pronađe i popravi oštećenja na pohranjenim informacijama u sekvencama.

Stupanj komplementarnosti između dva lanca nukleinskih kiselina može varirati, od potpune komplementarnosti (svaki nukleotid je preko puta svoje suprotnosti) do nikakve komplementarnosti (svaki nukleotid nije preko puta svoje suprotnosti) i određuje stabilnost sekvenci koje će biti zajedno. Nadalje, različite funkcije popravljanja DNK, kao i regulatorne funkcije, temelje se na komplementarnosti baznih parova. U biotehnologiji, princip komplementarnosti baznih parova omogućava stvaranje hibrida DNK, između RNK i DNK i otvara vrata modernim alatima kao što je biblioteka cDNK.
Iako se većina komplementarnosti vidi između dva odvojena niza nukleotida u DNK ili RNK, moguće je i da sekvenca ima unutrašnju komplementarnost što rezultira sekvencom vezanja za sebe u presavijenoj konfiguraciji.
Komplementarnost parova baza DNK i RNK
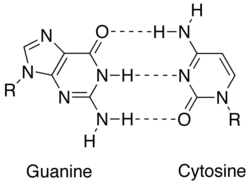
urediKomplementarnost se postiže različitim interakcijama između nukleobaza: adenin, timin (uracil u RNK), guanin i citozin. Adenin i guanin su purini, dok su timin, citozin i uracil pirimidini. Purini su veći od pirimidina. Oba tipa molekula se međusobno nadopunjuju i mogu se bazirati samo u paru s suprotnim tipom nukleobaze. U nukleinskoj kiselini, nukleobaze se drže zajedno pomoću vodikove veze, koja efikasno djeluje samo između adenina i timina i između guanina i citozina. Osnovni komplement A = T dijeli dvije vodikove veze, dok bazni par G≡C ima tri takve veze. Sve ostale konfiguracije između nukleobaza ometale bi stvaranje dvostruke zavojnice. Lanci nukleotida u DNK orijentirani su u suprotnim smjerovima, pa se označavaju kao antiparalelni.[1]
| Nukleinska kiselina | Nukleobaza | Bazni komplement |
| DNK | Adenin (A), Timin (T), Guanin (G), Citozin (C) | A=T, G≡C |
| RNK | Adenin (A), Uracil (U), Guanin (G), Citozin (C) | A=U, G≡C |
Komplementarni lanac DNK ili RNA može se konstruirati na osnovu komplementarnosti nukleobaza.[2] Svaki osnovni par, A = T nasuprot G≡C, zauzima otprilike isti prostor, omogućavajući tako uvijenu dvostruku spiralu DNK bez ikakvih prostornih izobličenja. Vodikova veza između nukleobaza također stabilizira dvostruku spiralu DNK.[3]
Komplementarnost lanaca DNK u dvostrukoj zavojnici omogućava upotrebu jednog lanca kao predloška/matrice/kalupa za konstrukciju drugog. Ovaj princip ima važnu ulogu u replikaciji DNK, postavljajući temelje nasljeđivanja, objašnjavajući kako se genetčke informacije mogu prenijeti na sljedeću generaciju. Komplementarnost se koristi i u transkripciji DNK, koja generira lanac RNK iz DNK predloška.[4] Pored toga, virus humane imunodeficijencije, jednolančani RNK virus, kodira RNK-zavisnu DNK polimerazu (reverzna transkriptaza) koja koristi komplementarnost za katalizu replikacije genoma. Obrnuta transkriptaza može se prebacivati između dvije roditeljske genomke RNK izborom kopije rekombinacija tokom replikacije.[5]
mehanizmi popravljanja DNK kao što je probno čitanje temelje se na komplementarnosti i omogućavaju korekciju grešaka tokom replikacije DNK, uklanjanjem neusklađenih nukleobaza.[1] Općenito, oštećenja na jednom lancu DNK mogu se popraviti uklanjanjem oštećenog dijela i njegovom zamjenom, korištenjem komplementarnosti za kopiranje podataka s drugog lanca, što se događa u procesima popravki neusklađenosti, popravki ekscizijenukleotida i popravki baznih ekscizija.[6]
Lanci nukleinskih kiselina mogu takođe formirati hibride u kojima jednolančana DNK može lahko sažeti sa komplementarnom DNK ili RNK. Ovaj princip je osnova uobičajenih laboratorijskih tehnika kao što je polimerazna lančana reakcija, PCR.[1]
Dva lanca komplementarne sekvence nazivaju se smisleni i antismisleni. Smisleni lanac je, općenito, transkribirana sekvenca DNK ili RNK, koja je generirana u transkripciji. Dok je antismisleni onaj koji je komplementaran smislenoj sekvenci
Samodopunjavanje i ukosničke petlje
urediSamokomplementarnost se odnosi na činjenicu da se sekvenca DNK ili RNK može saviti unatrag, stvarajući dvolančanu strukturu. Ovisno o tome koliko su dijelovi sekvenci međusobno komplementarni, lanac ože oblikovati ukosnice, spojeve, izbočine ili unutrašnje petlje.[1] Za RNK je vjerojatnije da će stvoriti ove vrste struktura zbog vevanja para baza koje se ne vide u DNK, poput veze guanina s uracilom.[1]
Regulacijske funkcije
urediKomplementarnost se može naći između kratkog istezanja nukleinske kiseline i kodirajućeg područja ili transkribiranog gena, a rezultira uparivanjem baza. Ove kratke sekvence nukleinskih kiselina obično se nalaze u prirodi i imaju regulacijske funkcije kao što je utišavanje gena.[1]
Antisensni transkripti
urediAntismisleni transkripti su dijelovi nekodirajuće iRNK, koji su komplementarni kodirajućoj sekvenci.[7] Studije širom genoma pokazale su da se u prirodi često javljaju antismisleni tralskripti RNK. Općenito se vjeruje da povećavaju potencijal kodiranja genetičke poruke i dodaju sveukupni sloj regulacije gena. Do sada je poznato da se 40% ljudskog genoma transkribira u oba smjera, naglašavajući potencijalni značaj reverzne transkripcije.[8]
Sugerira se da bi komplementarne regije između smislenih i antismislenih transkripata omogućile stvaranje dvolančanih hibrida RNK, koji mogu imati važnu ulogu u regulaciji gena. Na primjer, faktor induciran hipoksijom 1α iRNK i β-sekretaza iRNK transkribiraju se dvosmjerno i pokazalo se da antisens transkript djeluje kao stabilizator smislenog zapisa.[9]
miRNK i siRNK
uredimiRNK , mikroRNK su kratke sekvence RNK, koje su komplementarne regijama transkribiranog gena i imaju regulatorne funkcije. Sadašnja istraživanja pokazuju da se cirkulirajuća miRNK može koristiti kao novi biomarker, pokazujući obećavajuće aktivnosti koje će se koristiti u dijagnostici bolesti.[10] MiRNK se formiraju iz dužih sekvenci RNK koje dicer enzim odsjeca iz sekvence RNK koja je iz regulacijskog gena. Te kratke niti vežu se za RISC kompleks. Oni se podudaraju sa sekvencama u uzvodnom području prepisanog gena, zbog njihove komplementarnosti da djeluju kao prigušivač gena na tri načina. Jedan je sprečavanje ribosoma da se veže i pokretanje translacije. Dva su putem degradacije iRNK na koju se kompleks vezao. A treći je pružanje nove sekvence dvolančane RNK (dsRNA) na koju može djelovati dicer, kako bi stvorio više miRNK i pronašao i razgradio više kopija gena. Male ometajuće molekule RNK (siRNK) slične su po funkciji miRNK; potiču iz drugih izvora RNK, ali imaju sličnu svrhu kao i miRNK.[1]
S obzirom na njihovu kratku dužinu, pravila komplementarnosti znače da i dalje mogu biti vrlo diskriminirajući u odabiru svojih ciljeva. Budući da postoje četiri izbora za svaku bazu u lancu i dužinu od 20 bp - 22bp za mi/siRNK, što dovodi do više od 1×1012 mogućih kombinacija. Kako je ljudski genom dugačak ~3,1 milijarde baza,[11] što znači da bi svaka miRNK trebala slučajno pronaći podudaranje samo jednom u čitavom ljudskom genomu.
Priljubljujuće ukosnice
urediUkosnice koje se priljubljuju nastaju kada se jedan lanac nukleinske kiseline nadopuni samim sobom stvarajući petlje RNA u obliku ukosnice.[12] Kada dvije ukosnice dođu u međusobni kontakt in vivo, komplementarne baze formiraju dviju niti i počinju odmatavati ukosnice dok se ne formira dvolančani RNK (dsRNK) kompleks ili se kompleks ne odmota natrag na dva odvojene sekvence, zbog neusklađenosti ukosnica. Sekundarna struktura ukosnice prije priljubljivanja omogućava stabilnu strukturu s relativno fiksnom promjenom energije.[13] Svrha ovih struktura je balansiranje stabilnosti petlje ukosnice u odnosu na čvrstoću vezanja s komplementarnom niti. Prejako početno vezanje za lošu lokaciju i sekvence neće se odviti dovoljno brzo; preslabo početno vezanje lanaca nikada neće u potpunosti formirati željeni kompleks. Ove strukture ukosnica omogućuju izlaganje dostatno baza da omoguće dovoljno snažnu provjeru početnog vezanja i dovoljno slabo unutrašnje vezivanje da omoguće odvijanje nakon što se pronađe povoljno podudaranje.[13]
---C G--- C G ---C G--- U A C G G C U A C G G C A G C G A A A G C U A A U CUU ---CCUGCAACUUAGGCAGG--- A GAA ---GGACGUUGAAUCCGUCC--- G A U U U U U C U C G C G C C G C G A U A U G C G C ---G C--- ---G C---
Priljubljivanje ukosnica na vrhu petlji. Komplementarnost od dvije glave podstiče ukosnicu da se otvori i poravna, postajući jedna ravna sekvenca od dvije niti, a ne dvije ukosnice.
Bioinformatika
urediKomplementarnost omogućava da se informacije pronađene u DNK ili RNK čuvaju u jednom lancu. Lanac dopunjavanja može se odrediti iz predloška i obrnuto, kao u cDNA bibliotekama. Ovo takođe omogućava analizu, poput upoređivanja sekvenci dviju različitih vrsta. Za zapisivanje sekvenci razvijene su skraćenice, kada postoje neusklađenosti (dvosmisleni kodovi) ili za ubrzavanje čitanja suprotnog niza u komplementu (ambigrami).
Biblioteka cDNK
urediBiblioteka cDNK je kolekcija izraženih gena DNK, koji se smatraju korisnim referentnim alatom u procesima identifikacije i kloniranja gena. Ove biblioteke su izrađene od iRNK, koristeći RNK-ovisnu reverznu transkriptazu DNK-polimeraze (RT), koja transkribira iRNK obrazac u DNK. Stoga biblioteka cDNK može sadržavati samo umetke koji su namijenjeni transkripciji u iRNK. Ovaj proces se oslanja na princip komplementarnosti DNK/ RNK. Krajnji proizvod biblioteka je dvolančana DNK koja se može umetnuti u plazmide. Stoga su biblioteke cDNK moćan alat u modernim istraživanjima.[1][14]
Dvosmisleni kodovi
urediKada se pišu sekvence za sistematsku biologiju možda će biti potrebni IUPAC kodovi koji znače "bilo koji od dva" ili "bilo koji od tri". IUPAC kod R (bilo koji purin) komplementaran je Y (bilo koji pirimidin) i M (amino) K (keto). W (slabo) i S (jako) obično se ne zamjenjuju.[15] ali su ih u prošlosti zamijenili neki alati.[16] W i S označavaju "slabe", odnosno "jake" i ukazuju na niz vodikovih veza koje u nukleotidu, za uparivanje sa svojim dopunjujućim parnjakom. Parnjak koristi isti broj veza za stvaranje komplementarnog para.[17]
IUPAC kod koji specifično isključuje jedan od tri nukleotida može biti komplementaran IUPAC kodu koji isključuje komplementarni nukleotid. Na primjer, V (A, C ili G - "nije T") može dopunjavati B (C, G ili T - "ne A").
| Simbol[18] | Opis | Prezentirane baze | ||||
|---|---|---|---|---|---|---|
| A | adenin | A | 1 | |||
| C | citozin | C | ||||
| G | guanin | G | ||||
| T | timin | T | ||||
| U | uracil | U | ||||
| W | weak = slabo | A | T | 2 | ||
| S | strong =jako | C | G | |||
| M | amino | A | C | |||
| K | keto | G | T | |||
| R | purin | A | G | |||
| Y | pirimidin | C | T | |||
| B | ne A (B ide nakon A) | C | G | T | 3 | |
| D | ne C (D ide nakon C) | A | G | T | ||
| H | ne G (H ide nakon G) | A | C | T | ||
| V | ne T (V ide nakon T i U) | A | C | G | ||
| N ili – | ani baza (ne gep) | A | C | G | T | 4 |
Ambigrami
urediOdređene oznake mogu se koristiti za stvaranje odgovarajućeg (ambigrafskog) označavanja za komplementarne baze nukleinske kiseline (tj. guanin = b, citozin = q, adenin = n i timin = u), što omogućava dopunu čitavih sekvenci DNK, jednostavnim rotiranjem teksta "naopako".[19] Naprimjer, s prethodnom abecedom, ;ubnq (GTCA) čitalo bi se kao
- ubnq (TGAC, obrnuti dodatak) ako se okrene naopako.
- qqubqnnquunbbqnbb
- bbnqbuubnnuqqbuqq
Ambigrafski zapisi lahko vizueliziraju komplementarne dijelove nukleinske kiseline, kao što su palindromske sekvence.[20] Ova funkcija je poboljšana kada se koriste prilagođeni fontovi ili simboli, a ne uobičajeni ASCII ili čak Unicode karakteri.[20]
Također pogledajte
urediReference
uredi- ^ a b c d e f g h Watson, James, Cold Spring Harbor Laboratory, Tania A. Baker, Massachusetts Institute of Technology, Stephen P. Bell, Massachusetts Institute of Technology, Alexander Gann, Cold Spring Harbor Laboratory, Michael Levine, University of California, Berkeley, Richard Losik, Harvard University ; with Stephen C. Harrison, Harvard Medical (2014). Molecular biology of the gene (Seventh izd.). Boston: Benjamin-Cummings Publishing Company. ISBN 978-0-32176243-6.
- ^ Pray, Leslie (2008). "Discovery of DNA structure and function: Watson and Crick". Nature Education. 1 (1): 100. Pristupljeno 27. 11. 2013.
- ^ Shankar, A; Jagota, A; Mittal, J (Oct 11, 2012). "DNA base dimers are stabilized by hydrogen-bonding interactions including non-Watson-Crick pairing near graphite surfaces". The Journal of Physical Chemistry B. 116 (40): 12088–94. doi:10.1021/jp304260t. PMID 22967176.
- ^ Hood, L; Galas, D (Jan 23, 2003). "The digital code of DNA". Nature. 421 (6921): 444–8. Bibcode:2003Natur.421..444H. doi:10.1038/nature01410. PMID 12540920.
- ^ Rawson JMO, Nikolaitchik OA, Keele BF, Pathak VK, Hu WS. Recombination is required for efficient HIV-1 replication and the maintenance of viral genome integrity. Nucleic Acids Res. 2018;46(20):10535-10545. DOI:10.1093/nar/gky910 PMID 30307534
- ^ Fleck O, Nielsen O. DNA repair. J Cell Sci. 2004;117(Pt 4):515-517. DOI:10.1242/jcs.00952
- ^ He, Y; Vogelstein, B; Velculescu, VE; Papadopoulos, N; Kinzler, KW (Dec 19, 2008). "The antisense transcriptomes of human cells". Science. 322 (5909): 1855–7. Bibcode:2008Sci...322.1855H. doi:10.1126/science.1163853. PMC 2824178. PMID 19056939.
- ^ Katayama, S; Tomaru, Y; Kasukawa, T; Waki, K; Nakanishi, M; Nakamura, M; Nishida, H; Yap, CC; Suzuki, M; Kawai, J; Suzuki, H; Carninci, P; Hayashizaki, Y; Wells, C; Frith, M; Ravasi, T; Pang, KC; Hallinan, J; Mattick, J; Hume, DA; Lipovich, L; Batalov, S; Engström, PG; Mizuno, Y; Faghihi, MA; Sandelin, A; Chalk, AM; Mottagui-Tabar, S; Liang, Z; Lenhard, B; Wahlestedt, C; RIKEN Genome Exploration Research Group; Genome Science Group (Genome Network Project Core Group); FANTOM Consortium (Sep 2, 2005). "Antisense transcription in the mammalian transcriptome". Science. 309 (5740): 1564–6. Bibcode:2005Sci...309.1564R. doi:10.1126/science.1112009. PMID 16141073.
- ^ Faghihi, MA; Zhang, M; Huang, J; Modarresi, F; Van der Brug, MP; Nalls, MA; Cookson, MR; St-Laurent G, 3rd; Wahlestedt, C (2010). "Evidence for natural antisense transcript-mediated inhibition of microRNA function". Genome Biology. 11 (5): R56. doi:10.1186/gb-2010-11-5-r56. PMC 2898074. PMID 20507594.
- ^ Kosaka, N; Yoshioka, Y; Hagiwara, K; Tominaga, N; Katsuda, T; Ochiya, T (Sep 5, 2013). "Trash or Treasure: extracellular microRNAs and cell-to-cell communication". Frontiers in Genetics. 4: 173. doi:10.3389/fgene.2013.00173. PMC 3763217. PMID 24046777.
- ^ "Ensembl genome browser 73: Homo sapiens - Assembly and Genebuild". Ensembl.org. Arhivirano s originala, 15. 2. 2013. Pristupljeno 27. 11. 2013.
- ^ Marino, JP; Gregorian RS, Jr; Csankovszki, G; Crothers, DM (Jun 9, 1995). "Bent helix formation between RNA hairpins with complementary loops". Science. 268 (5216): 1448–54. Bibcode:1995Sci...268.1448M. doi:10.1126/science.7539549. PMID 7539549.
- ^ a b Chang, KY; Tinoco I, Jr (30. 5. 1997). "The structure of an RNA "kissing" hairpin complex of the HIV TAR hairpin loop and its complement". Journal of Molecular Biology. 269 (1): 52–66. doi:10.1006/jmbi.1997.1021. PMID 9193000.
- ^ Wan, KH; Yu, C; George, RA; Carlson, JW; Hoskins, RA; Svirskas, R; Stapleton, M; Celniker, SE (2006). "High-throughput plasmid cDNA library screening". Nature Protocols. 1 (2): 624–32. doi:10.1038/nprot.2006.90. PMID 17406289.
- ^ Jeremiah Faith (2011), conversion table
- ^ arep.med.harvard.edu A tool page with the note about the applied W-S conversion patch.
- ^ Reverse-complement tool page with documented IUPAC code conversion, source code available.
- ^ Nomenclature Committee of the International Union of Biochemistry (NC-IUB) (1984). "Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences". Pristupljeno 4. 2. 2008.
- ^ Rozak DA (2006). "The practical and pedagogical advantages of an ambigraphic nucleic acid notation". Nucleosides Nucleotides Nucleic Acids. 25 (7): 807–13. doi:10.1080/15257770600726109. PMID 16898419.
- ^ a b Rozak, DA; Rozak, AJ (maj 2008). "Simplicity, function, and legibility in an enhanced ambigraphic nucleic acid notation". BioTechniques. 44 (6): 811–3. doi:10.2144/000112727. PMID 18476835.