trp operon
trp operon je grupa gena koji se transkribiraju zajedno, kodirajući enzime koji u bakterijama proizvode aminokiselinu triptofan. trp operon je prvi put okarakterisan u Escherichia coli, a od tada je otkriven u mnogim drugim bakterijama.[1] Operon je reguliran tako da, kada je triptofan prisutan u okolini, geni za sintezu triptofana su potisnuti.
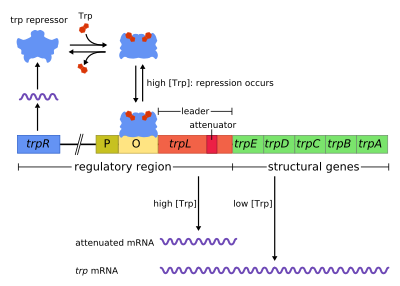
"trp" operon sadrži pet strukturnih gena: trpE, trpD, trpC, trpB i trpA, koji kodiraju enzime potrebne za sintezu triptofana. Također sadrži represivni regulatorski gen koji se zove trpR. Kada je prisutan triptofan, protein trpR se vezuje za operator, blokirajući transkripciju trp operona pomoću RNK-polimeraze.
Ovaj operon je primjer represivne negativne regulacije ekspresije gena. Represorski protein vezuje se za operator u prisustvu triptofana (potiskujući transkripciju) i oslobađa se iz operona kada je triptofan odsutan (omogućava transkripcija za nastavak). Operaon trp dodatno koristi slabljenje za kontrolu ekspresije operona, drugog kontrolnog mehanizma negativne povratne sprege.
trp operon je dobro proučen i obično se koristi kao primjer regulacije gena u bakterijama uz lac operon.
Geni
urediTrp operon sadrži pet strukturnih gena. Njihove uloge su:
- TrpE (P00895): Antranilat-sintaza proizvodi antranilat.
- TrpD (P00904): Sarađuje sa TrpE.
- TrpC (P00909): Fosforibozilantranilat-izomeraza domen prvo pretvara N-(5-fosfo-β-D-ribozil)antranilat u 1-(2-karboksifenilamino)-1-deoksi-D- ribuloza 5-fosfat. Indol-3-glicerol-fosfat sintaza na istom proteinu zatim pretvara proizvod u (1S,2R)-1-C-(indol-3-il)glicerol 3-fosfat.
- TrpA (P0A877), TrpB (P0A879): dvije podjedinice triptofan sintetaze. Kombinira TrpC-ov proizvod sa serinom za proizvodnju triptofana.
Represija
urediOperon djeluje putem mehanizma negativne povratne sprege. Represor za trp operon proizvodi se uzvodno od strane trpR gena, koji je konstitutivno eksprimiran na niskom nivou. Sintetizirani trpR monomeri povezuju se u dimere. Kada je triptofan prisutan, ovi triptofanski represorni dimeri vezuju se za triptofan, uzrokujući promjenu konformacije represora, omogućavajući represoru da se veže za operator. Ovo sprečava RNK-polimerazu da se veže i transkribuje operon, tako da se triptofan ne proizvodi iz njegovog prekursora. Kada triptofan nije prisutan, represor je u svojoj neaktivnoj konformaciji i ne može da veže region operatora, tako da represor ne inhibira transkripciju.
Atenuacija
urediPrigušivanje je drugi mehanizam negativne povratne sprege u trp operonu. Sistem represije cilja na unutarćelijsku koncentraciju trp-a, dok slabljenje reaguje na koncentraciju nabijene tRNKtrp.[2] Dakle, trpR represor smanjuje ekspresiju gena mijenjajući inicijaciju transkripcije, dok atenuacija to čini mijenjanjem procesa transkripcije koji je već u toku.[2] Dok TrpR represor smanjuje transkripciju za faktor od 70 , slabljenje ga može dalje smanjiti za faktor od 10, čime se omogućava akumulirana represija od oko 700 puta.[3] Slabljenje je omogućeno činjenicom da u prokariotima (koji nemaju jedro), ribosomi počinju prevođenje iRNK dok RNK-polimeraza još uvijek transkribuje sekvencu DNK. Ovo omogućava da proces prevođenja direktno utiče na transkripciju operona.
Na početku transkribovanih gena trp operona nalazi se sekvenca od najmanje 130 nukleotida koji se naziva vodeći transkript (trpL; P0AD92).[4] Lee i Yanofsky (1977) otkrili su da je efikasnost atenuacije u korelaciji sa stabilnošću sekundarne strukture ugrađene u trpL,[5] a 2 sastavne ukosničke strukture terminatora kasnije su razjasnili Oxender et al. (1979).[6] Ovaj transkript uključuje četiri kratke sekvence označene sa 1–4, od kojih je svaka djelimično komplementarna sljedećoj. Tako se mogu formirati tri različite sekundarne strukture (ukosnice): 1–2, 2–3 ili 3–4. Hibridizacija sekvenci 1 i 2, kako bi se formirala struktura 1-2 je rijetka jer RNK-polimeraza čeka da se ribosom pričvrsti prije nego što nastavi transkripciju nakon sekvence 1; međutim, ako bi se formirala ukosnica 1–2, spriječila bi stvaranje 2–3 struktura (ali ne 3–4). Formiranje ukosnice između sekvenci 2–3 sprečava stvaranje ukosnica između 1–2 i 3–4. Struktura 3–4 je transkripcijskaa terminacijska sekvenca (obiluje G/C i odmah praćena sa nekoliko ostataka uracila), nakon što se formira, RNK-polimeraza će se odvojiti od DNK i transkripcije strukturnih gena operona se ne mogu pojaviti (vidi dolje za detaljnije objašnjenje). Funkcionalni značaj 2. ukosnice za terminaciju transkripcije ilustruje smanjena frekvencija terminacije transkripcije uočena u eksperimentima koji destabilizuju centralno G+C uparivanje ove ukosnice.[5][7][8][9]
Dio vodećeg transkripta kodira kratki polipeptid od 14 aminokiselina, zvani vodeći peptid. Ovaj peptid sadrži dva susjedna ostatka triptofana, što je neobično, budući da je triptofan prilično neuobičajena aminokiselina (otprilike jedan od stotinu ostataka u tipskom proteinu "E. coli" je triptofan). Lanac 1 u trpL obuhvata regiju koja kodira zadnje ostatke vodećeg peptida: Trp, Trp, Arg, Thr, Ser;[2] uočena je konzerviranost u ovih 5 kodona, dok mutiranje uzvodnih kodona nije promijenilo ekspresiju operona.[2][10][11][12] Ako ribosom pokuša da prevede ovaj peptid dok su nivoi triptofana u ćeliji niski, on će se zaustaviti na jednom od dva trp kodona. Dok je u zastoju, ribosom fizički štiti sekvencu transkripta1, sprečavajući formiranje 1–2 sekundarne strukture. Sekvenca 2 se tada može slobodno hibridizirati sa sekvencom 3 kako bi formirala strukturu 2–3, koja tada sprječava formiranje 3–4 terminacijske ukosnice, zbog čega se struktura 2–3 naziva antiterminacijskom ukosnicom. U prisustvu strukture 2–3, RNK-polimeraza je slobodna da nastavi sa transkripcijom operona. Mutacijska analiza i studije koje uključuju komplementarne oligonukleotide pokazuju da stabilnost strukture 2–3 odgovara nivou ekspresije operona l.[10][13][14][15] Ako su nivoi triptofana u ćeliji visoki, ribosom će prevesti ceo vodeći peptid bez prekida i samo će se zaustaviti tokom završetka translacije na stop kodonu. U ovom trenutku ribosom fizički štiti obje sekvence, i 1 i 2. Sekvence 3 i 4 su stoga slobodne da formiraju strukturu 3–4 koja završava transkripciju. Ova struktura terminatora formira se kada nijedan ribosom ne stane u blizini Trp tandema (tj. Trp ili Arg kodona): ili vodeći peptid nije preveden ili se translacija odvija glatko duž lanca 1 sa obilnim nabijenim tRNKtrp.[10][2] Štaviše, predlaže se da ribosom blokira samo oko 10 nt nizvodno, tako da ribosom zastaje u uzvodnom Gly ili dalje nizvodnom Thr ne utiče na formiranje završne ukosnice.[2][10] Krajnji rezultat je da će operon biti transkribovan samo kada triptofan nije dostupan za ribosom, dok je trpL transkript konstitutivno izražen.
Ovaj mehanizam atenuacije je eksperimentalno podržan. Prvo, direktno je dokazano da su translacija vodećeg peptida i zastoj ribosoma neophodni za inhibiciju terminacije transkripcije.[13] Štaviše, analiza mutacije koja destabilizuje ili ometa uparivanje baza antiterminatorske ukosnice dovodi do povećanog završetka nekoliko nabora; u skladu s modelom atenuacije, ova mutacija ne uspijeva ublažiti slabljenje čak i sa izgladnjelim Trp.[10][13] Nasuprot tome, komplementarni oligonukleotidi koji ciljaju lanac 1 povećavaju ekspresiju operona podsticanjem formiranje antiterminatora.[10][14] Nadalje, u histidinskom operonu, kompenzacijska mutacija pokazuje da je sposobnost uparivanja niti 2–3 važnija od njihove primarne sekvence u inhibiciji slabljenja.[10][15]
U atenuaciji, gdje se translacijski ribosom zaustavlja, određuje da li će se formirati ukosnica za završetak.[10] Da bi transkribirajuća polimeraza istovremeno uhvatila alternativnu strukturu, vremenska skala strukturne modulacije mora biti uporediva na transkripciju.[2] Da bi se osiguralo da se ribosom veže i započne translaciju vodećeg transkripta odmah nakon njegove sinteze, u trpL sekvenci postoji mjesto za pauzu. Kada stigne na ovo mjesto, RNK-polimeraza pauzira transkripciju i očigledno čeka da počne translacija. Ovaj mehanizam omogućava sinhronizaciju transkripcije i translacije, što je ključni element u atenuaciji.
Sličan mehanizam slabljenja (atenuacije) reguliše sintezu histidina, fenilalanina i treonina.
Regulacija trp operona u Bacillus subtilis
urediTrp operonski geni raspoređeni su istim redoslijedom u E. coli i Bacillus subtilis.[16] Regulacija trp operona u oba organizma zavisi od količine trp u ćeliji. Međutim, primarna regulacija biosinteze triptofana u B. subtilis je putem atenuacije, a ne potiskivanja, transkripcije.[17] U B. subtilis, triptofan se vezuje za jedanaest podjedinica triptofanom aktiviranog RNK-vezujućeg proteina atenuacije (TRAP), koji aktivira sposobnost TRAP-a da se veže za trp vodeću RNK.[18][19] Vezivanje trp-aktiviranog TRAP-a za vodeću RNK rezultira formiranjem terminatorske strukture koja uzrokuje završetak transkripcije.[17]
Reference
uredi- ^ Merino, Enrique; Jensen, Roy A; Yanofsky, Charles (april 2008). "Evolution of bacterial trp operons and their regulation". Current Opinion in Microbiology. 11 (2): 78–86. doi:10.1016/j.mib.2008.02.005. ISSN 1369-5274. PMC 2387123. PMID 18374625.
- ^ a b c d e f g Yanofsky C (februar 1981). "Attenuation in the control of expression of bacterial operons". Nature. 289 (5800): 751–8. Bibcode:1981Natur.289..751Y. doi:10.1038/289751a0. PMID 7007895. S2CID 4364204.
- ^ Lehninger AL, Nelson DL, Cox MM (2008). Principles of Biochemistry (5th izd.). New York, NY: W.H. Freeman and Company. str. 1128. ISBN 978-0-7167-7108-1.
- ^ Bertrand K, Squires C, Yanofsky C (maj 1976). "Transcription termination in vivo in the leader region of the tryptophan operon of Escherichia coli". Journal of Molecular Biology. 103 (2): 319–37. doi:10.1016/0022-2836(76)90315-6. PMID 781269.
- ^ a b Lee F, Yanofsky C (oktobar 1977). "Transcription termination at the trp operon attenuators of Escherichia coli and Salmonella typhimurium: RNA secondary structure and regulation of termination". Proceedings of the National Academy of Sciences of the United States of America. 74 (10): 4365–9. Bibcode:1977PNAS...74.4365L. doi:10.1073/pnas.74.10.4365. PMC 431942. PMID 337297.
- ^ Oxender DL, Zurawski G, Yanofsky C (novembar 1979). "Attenuation in the Escherichia coli tryptophan operon: role of RNA secondary structure involving the tryptophan codon region". Proceedings of the National Academy of Sciences of the United States of America. 76 (11): 5524–8. Bibcode:1979PNAS...76.5524O. doi:10.1073/pnas.76.11.5524. PMC 411681. PMID 118451.
- ^ Stroynowski I, Yanofsky C (juli 1982). "Transcript secondary structures regulate transcription termination at the attenuator of S. marcescens tryptophan operon". Nature. 298 (5869): 34–8. Bibcode:1982Natur.298...34S. doi:10.1038/298034a0. PMID 7045685. S2CID 4347442.
- ^ Zurawski G, Yanofsky C (septembar 1980). "Escherichia coli tryptophan operon leader mutations, which relieve transcription termination, are cis-dominant to trp leader mutations, which increase transcription termination". Journal of Molecular Biology. 142 (1): 123–9. doi:10.1016/0022-2836(80)90210-7. PMID 6159477.
- ^ Stauffer GV, Zurawski G, Yanofsky C (oktobar 1978). "Single base-pair alterations in the Escherichia coli trp operon leader region that relieve transcription termination at the trp attenuator". Proceedings of the National Academy of Sciences of the United States of America. 75 (10): 4833–7. Bibcode:1978PNAS...75.4833S. doi:10.1073/pnas.75.10.4833. PMC 336215. PMID 368800.
- ^ a b c d e f g h Kolter R, Yanofsky C (1982). "Attenuation in amino acid biosynthetic operons". Annual Review of Genetics. 16: 113–34. doi:10.1146/annurev.ge.16.120182.000553. PMID 6186194.
- ^ Lee F, Bertrand K, Bennett G, Yanofsky C (maj 1978). "Comparison of the nucleotide sequences of the initial transcribed regions of the tryptophan operons of Escherichia coli and Salmonella typhimurium". Journal of Molecular Biology. 121 (2): 193–217. doi:10.1016/s0022-2836(78)80005-9. PMID 351195.
- ^ Miozzari G, Yanofsky C (novembar 1978). "Naturally occurring promoter down mutation: nucleotide sequence of the trp promoter/operator/leader region of Shigella dysenteriae 16". Proceedings of the National Academy of Sciences of the United States of America. 75 (11): 5580–4. Bibcode:1978PNAS...75.5580M. doi:10.1073/pnas.75.11.5580. PMC 393010. PMID 364484.
- ^ a b c Zurawski G, Elseviers D, Stauffer GV, Yanofsky C (decembar 1978). "Translational control of transcription termination at the attenuator of the Escherichia coli tryptophan operon". Proceedings of the National Academy of Sciences of the United States of America. 75 (12): 5988–92. Bibcode:1978PNAS...75.5988Z. doi:10.1073/pnas.75.12.5988. PMC 393102. PMID 366606.
- ^ a b Winkler ME, Mullis K, Barnett J, Stroynowski I, Yanofsky C (april 1982). "Transcription termination at the tryptophan operon attenuator is decreased in vitro by an oligomer complementary to a segment of the leader transcript". Proceedings of the National Academy of Sciences of the United States of America. 79 (7): 2181–5. Bibcode:1982PNAS...79.2181W. doi:10.1073/pnas.79.7.2181. PMC 346154. PMID 6179092.
- ^ a b Johnston HM, Roth JR (februar 1981). "DNA sequence changes of mutations altering attenuation control of the histidine operon of Salmonella typhimurium". Journal of Molecular Biology. 145 (4): 735–56. doi:10.1016/0022-2836(81)90312-0. PMID 6167727.
- ^ Merino E, Jensen RA, Yanofsky C (april 2008). "Evolution of bacterial trp operons and their regulation". Current Opinion in Microbiology. 11 (2): 78–86. doi:10.1016/j.mib.2008.02.005. PMC 2387123. PMID 18374625.
- ^ a b Gollnick P, Babitzke P, Antson A, Yanofsky C (14. 11. 2005). "Complexity in regulation of tryptophan biosynthesis in Bacillus subtilis". Annual Review of Genetics. 39 (1): 47–68. doi:10.1146/annurev.genet.39.073003.093745. PMID 16285852.
- ^ Elliott MB, Gottlieb PA, Gollnick P (januar 2001). "The mechanism of RNA binding to TRAP: initiation and cooperative interactions". RNA. 7 (1): 85–93. doi:10.1017/S135583820100173X. PMC 1370072. PMID 11214184.
- ^ Antson AA, Otridge J, Brzozowski AM, Dodson EJ, Dodson GG, Wilson KS, et al. (april 1995). "The structure of trp RNA-binding attenuation protein". Nature. 374 (6524): 693–700. Bibcode:1995Natur.374..693A. doi:10.1038/374693a0. PMID 7715723. S2CID 4340136.
Dopunska literatura
uredi- Morse DE, Mosteller RD, Yanofsky C (1969). "Dynamics of synthesis, translation, and degradation of trp operon messenger RNA in E. coli". Cold Spring Harbor Symposia on Quantitative Biology. 34: 725–40. doi:10.1101/sqb.1969.034.01.082. PMID 4909527.
- Yanofsky C (februar 1981). "Attenuation in the control of expression of bacterial operons". Nature. 289 (5800): 751–8. Bibcode:1981Natur.289..751Y. doi:10.1038/289751a0. PMID 7007895. S2CID 4364204.
Vanjski linkovi
uredi