Popravak neusklađenosti DNK
Popravak neusklađenosti je specifičan za niti. Tokom sinteze DNK, novosintetizovani (kćerinski) lanac obično uključuje greške. Da bi se započela popravka, mehanizam za popravak neusklađenosti razlikuje novosintetizirani lanac od šablona (roditeljskog). Kod gram-negativnih bakterija, prolazna hemimetilacija razlikuje niti (roditeljska je metilirana, a kćerinska nije). Međutim, kod drugih prokariota i eukariota, tačan mehanizam nije jasan. Sumnja se da, kod eukariota, novosintetizovana DNK sa zaostalim lancem prolazno sadrži ureze (prije nego što bude zapečaćena DNK ligazom) i daje signal koji usmjerava neusklađene sisteme korekture na odgovarajući lanac. To implicira da ovi urrezi moraju biti prisutni u vodećim niti, a dokazi za to su nedavno pronađeni[3] Recent work[4] pokazuju da su urezi mjesta za RFC-ovisno učitavanje klizne spone za replikaciju, proliferirajući ćelijski jedarni antigen (PCNA), na način specifičan za orijentaciju, tako da je jedno lice proteina u obliku krofne suprotstavljeno prema kraju 3'-OH na urezu. Opterećena PCNA zatim usmjerava djelovanje MutLalpha endonukleaze [5] lancu kćeri u prisustvu neusklađenosti i MutSalpha ili MutSbeta.
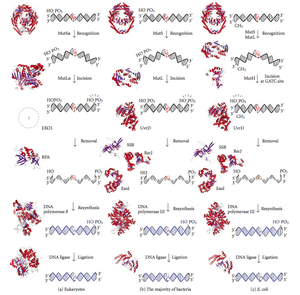
- Popravak neusklađenosti DNK (eng. Mismatch repair ; MMR) je sistem za prepoznavanje i popravljanje pogrešnih inesrcija, delecija i pogrešnog ugradnje baza koje može nastati tokom DNK replikacija i rekombinacija, kao i popravka nekih oblika oštećenja DNK.[1][2]
Svaki mutacijski događaj koji poremeti superheliksnu strukturu DNK nosi sa sobom potencijal da ugrozi genetičku stabilnost ćelije. Činjenica da su sistemi za otkrivanje oštećenja i popravke složeni koliko i sam mehanizam za replikaciju naglašava važnost koju je evolucija pridavala vjernosti strukture DNK.
Primjeri neusklađenih baza uključuju G/T ili A/C uparivanje (pogledajte popravak DNK). Nepodudarnosti su obično uzrokovane tautomerizacijom baza tokom replikacije DNK. Oštećenje se popravlja prepoznavanjem deformiteta uzrokovanog neusklađenošću, određivanjem predloška i lanca koji nije predložak, te izrezivanjem pogrešno ugrađene baze i zamjenom ispravnim nukleotidom. Proces uklanjanja uključuje više od samog neusklađenog nukleotida. Može se ukloniti nekoliko ili čak do hiljada parova baza novosintetizovanog lanca DNK.
Proteini za popravku neusklađenosti
uredi| Protein za popravku neusklađenosti DNK, C-terminalni domen | |
|---|---|
| hpms2-atpgs | |
| Identifikatori | |
| Simbol | DNK-mis-repair |
Popravak neusklađenosti je visoko konzerviran proces od prokariota do eukariota. Prvi dokaz za popravku neusklađenosti dobijen je od S. pneumoniae (hexA i hexB gena). Naknadni rad na E. coli je identifikovao niz gena koji, kada se mutacijom inaktiviraju, uzrokuju hipermutabilne sojeve. Genetički proizvodi se, stoga, nazivaju "Mut" proteini i glavne su aktivne komponente sistema za popravku neusklađenosti. Tri od ovih proteina su neophodna u otkrivanju neusklađenosti i usmjeravanju mehanizma za popravku na njega: MutS, MutH i MutL (MutS je homolog HexA i MutL HexB).
MutS formira dimer (MutS2) koji prepoznaje neusklađenu bazu na lancu kćeri i vezuje mutiranu DNK. MutH se veže na hemimetiliranim mjestima duž ćerke DNK, ali je njegovo djelovanje latentno, aktivira se tek nakon kontakta s MutL dimerom (MutL2), koji vezuje MutS-DNK kompleks i djeluje kao posrednik između MutS2 i MutH, aktiviranje potonjeg. DNK se izvlači u petlju kako bi se tražilo najbliže d(GATC) metilacijsko mjesto neusklađenosti, koje bi moglo biti udaljeno do 1 kb. Nakon aktivacije kompleksom MutS-DNK, MutH odsijeca lanac kćer blizu hemimetiliranog mjesta. MutL regrutuje UvrD helikaza (DNK helikaza II) da razdvoji dva lanca sa specifičnim polaritetom od 3' do 5'. Cijeli MutSHL kompleks tada klizi duž DNK u smjeru nepodudaranja, oslobađajući lanac koji treba biti izrezan dok ide. Egzonukleaza prati kompleks i probavlja rep ss-DNK. Regrutovana egzonukleaza zavisi od toga na kojoj strani nepodudaranja MutH urezuje lanac – 5' ili 3'. Ako je urez koji je napravio MutH na 5' kraju neslaganja, koristi se ili RecJ ili ExoVII (oba 5' do 3' egzonukleaze). Međutim, ako je nadimak na 3' kraju neslaganja, koristi se egzoI (enzim od 3' do 5').
Čitav proces se završava nakon mjesta neslaganja - to jest, i samo mjesto i okolni nukleotidi su potpuno izrezani. Jednolančani jaz stvoren egzonukleazom može se zatim popraviti pomoću DNK-polimeraze III (uz pomoć proteina koji se vezuje za jedan lanac), koji koristi drugi lanac kao šablon, i konačno zapečaćen DNK ligazom. DNK-metilaza zatim brzo metilira kćerinski lanac.
MutS homolozi
urediU eukariotima, MutS homolozi formiraju dva glavna heterodimera: Msh2/Msh6 (MutSα) i Msh2/Msh3 (MutSβ). MutSα put je prvenstveno uključen u supstituciju baze i popravku neusklađenosti male petlje. Put MutSβ također je uključen u popravku male petlje, pored popravke velike petlje (~10 nukleotidnih petlji). Međutim, MutSβ ne popravlja zamjene baza.
MutL homolozi
urediMutL takođe ima slabu aktivnost ATPaza (koristi ATP za potrebe kretanja). Formira kompleks sa MutS i MutH, povećavajući otisak MutS-a na DNK.
Međutim, procesnost (udaljenost na kojoj enzim može da se kreće duž DNK prije disociacije) UvrD je samo ~40-50 bp. Budući da udaljenost između ureza stvorenog MutH-om i neusklađenosti može u prosjeku ~600 bp, ako nije napunjen drugi UvrD, odmotani dio je tada slobodan da se ponovo uklapa u svoj komplementarni lanac, prisiljavajući proces da počne iznova. Međutim, uz asistenciju MutL-a, "stopa" UvrD opterećenja se znatno povećava. Dok procesnost (i upotreba ATP-a) pojedinačnih UvrD molekula ostaje ista, ukupni efekat na DNK je značajno pojačan; DNK nema šanse da se ponovo zamota, jer svaki UvrD odmota 40-50 bp DNK, disocira, a zatim je odmah zamijenjen drugim UvrD, ponavljajući proces. Ovo izlaže velike dijelove DNK probavi egzonukleazama, što omogućava brzo izrezivanje (i kasniju zamjenu) netačne DNK.
Eukarioti imaju pet MutL homologa označenih kao MLH1, MLH2, MLH3, PMS1 i PMS2. Oni formiraju heterodimere koji oponašaju MutL u E. coli. Ljudski homolozi prokariotskog MutL formiraju tri kompleksa koji se nazivaju MutLα, MutLβ i MutLγ. MutLα kompleks je napravljen od MLH1 i PMS2 podjedinica, MutLβ heterodimer je napravljen od MLH1 i PMS1, dok je MutLγ napravljen od MLH1 i MLH3. MutLα djeluje kao endonukleaza koja uvodi prekide lanca u lanac kćeri nakon aktivacije neusklađenošću i drugim potrebnim proteinima, MutSα i PCNA. Ovi prekidi lanca služe kao ulazne tačke za aktivnost egzonukleaze koja uklanja neusklađenu DNK. Uloge koje igraju MutLβ i MutLγ u popravljanju neusklađenosti su manje shvaćene.
MutH je vrlo slaba endonukleaza koja se aktivira kada se veže za MutL (koji je i sam vezan za MutS). Ods9ieca nemetiliranu DNK i nemetilirani lanac hemimetilirane DNK, ali ne urezuje potpuno metiliranu DNK. Eksperimenti su pokazali da je popravak neusklađenosti slučajan ako nijedan lanac nije metiliran. Ova ponašanja su dovela do prijedloga da MutH određuje koja sekvenca sadrži neusklađenost. MutH nema eukariotski homolog. Njegovu endonukleaznu funkciju preuzimaju MutL homolozi, koji imaju specijaliziranu aktivnost 5'-3' egzonukleaze. Pristranost lanca za uklanjanje neusklađenosti iz novosintetizovane kćerke lanca kod eukariota može biti obezbijeđena slobodnim 3' krajevima Okazaki fragmenta u novom lancu stvorenom tokom replikacije.
PCNA β-klizna stezaljka
urediPCNA i β-klizna stezaljka povezana je sa MutSα/β i MutS, respektivno. Iako su prvi izvještaji sugerirali da PCNA-MutSα kompleks može poboljšati prepoznavanje neusklađenosti,[6] nedavno je pokazano [7] > da nema očigledne promjene u afinitetu MutSα za nepodudarnost u prisustvu ili odsustvu PCNA. Štaviše, mutanti MutSα koji nisu u stanju da stupe u interakciju sa PCNA in vitro pokazuju kapacitet da sprovedu prepoznavanje neusklađenosti i eksciziju neusklađenosti do nivoa blizu divljeg tipa. Takvi mutanti su defektni u reakciji popravljanja usmjerenoj prekidom lanca od 5', što sugerira po prvi put funkciju MutSα u koraku reakcije nakon ekscizije.
Klinički značaj
urediNaslijeđeni defekti u popravci neusklađenosti
urediMutacije u ljudskim homolozima Mut proteina utiču na stabilnost genoma, što može dovesti do mikrosatelitske nestabilnosti (MSI), uključene u neke tipove raka kod ljudi. Konkretno, nasljedni nepolipni kolorektumski karcinomi (HNPCC ili Lynchov sindrom) pripisuju se štetnim varijantama zametne linije u genima koji kodiraju MutS i MutL homologe MSH2 i MLH1, koji su tako klasifikovani kao tumor supresorski geni. Jedan podtip HNPCC, Muir-Toreovog sindroma (MTS), povezan je s tumorima kože. Ako obje naslijeđene kopije (alela) MMR gena nose štetne genske varijante, to rezultira vrlo rijetkim i teškim stanjem: sindrom raka popravljanja neusklađenosti (ili nedostatak konstitucijske popravke neslaganja, CMMR-D), koji se manifestira kao višestruko pojave tumora u ranoj dobi, često debelog crijeva i tumor mozga.[8]
Epigenetičko utišavanje gena za popravku neusklađenosti
urediSporadični karcinomi sa nedostatkom popravke DNK samo rijetko imaju mutaciju gena za popravak DNK, ali umjesto toga imaju tendenciju da imaju epigenetičke promjene kao što je metilacija promotora koja inhibira ekspresiju gena za popravak DNK.[9] Oko 13% kolorektumskih karcinoma ima nedostatak u popravci neusklađenosti DNK, obično zbog gubitka MLH1 (9,8%), ili ponekad MSH2, MSH6 ili PMS2 (svi ≤1.5%).[10] Za većinu sporadičnih kolorektumskih karcinoma sa nedostatkom MLH1, nedostatak je nastao zbog metilacije promotora MLH1.[10] Ostali tipovi karcinoma imaju veću učestalost gubitka MLH1 (pogledajte tabelu ispod), što je opet u velikoj mjeri posljedica metilacija promotora gena MLH1. Različiti epigenetički mehanizam koji leži u osnovi nedostataka MMR može uključivati prekomjernu ekspresiju mikroRNK, naprimjer miR-155 nivoi u obrnutoj korelaciji sa ekspresijom MLH1 ili MSH2 kod kolorektumskog karcinoma.[11]
| Tip raka | Učestalost deficita kod raka | Učestalost nedostataka u susjednom defektu polja |
|---|---|---|
| 32%[12][13] | 24%-28% | |
| Želudac (tumori foveolskog tipa) | 74%[14] | 71% |
| Želudac u dolini Kašmira visoke incidencije | 73%[15] | 20% |
| Jednjački | 73%[16] | 27% |
| Karcinom skvamoznih ćelija glave i vrata (HNSCC) | 31%-33%[17][18] | 20%-25% |
| Karcinom pluća nemalih ćelija (NSCLC) | 69%[19] | 72% |
| Kolorektumski | 10%[10] |
MMR kvarovi u defektima na terenu
uredidefekt polja (kancerizacija polja) je područje epitela koje je preduslovljeno epigenetičkim ili genetičkim promjenama, što ga predisponira za razvoj raka. Kako je istakao Rubin "...postoje dokazi da se više od 80% somatskih mutacija pronađenih u mutatorskom fenotipu ljudskih kolorektumsih tumora javlja prije početka terminalne klonske ekspanzije."[20][21] Slično tome, Vogelstein et al.[22] ističu da se više od polovine somatskih mutacija identifikovanih u tumorima dogodilo u preneoplazijskoj fazi (u defektu polja), tokom rasta naizgled normalnih ćelija.
Nedostaci MLH1 bili su česti kod defekta polja (histološki normalnog tkiva) koji okružuju tumore; vidi gornju tabelu. Epigenetički utišani ili mutirani MLH1 vjerovatno ne bi dao selektivnu prednost matičnoj ćeliji, međutim, to bi izazvalo povećane stope mutacija, a jedan ili više mutiranih gena može dati ćeliji selektivnu prednost. Deficitarni gen MLH1 bi se tada mogao nositi kao selektivno skoro neutralan putnički (autostoperski) gen kada mutirana matična ćelija generira prošireni klon. Kontinuirano prisustvo klona s epigenetićki potisnutim MLH1 nastavilo bi stvarati daljnje mutacije, od kojih bi neke mogle proizvesti tumor.
MMR komponente kod ljudi
urediKod ljudi, sedam proteina popravljanja neusklađenosti DNK (MMR) proteina (MLH1, MLH3, MSH2, MSH3, MSH6, PMS1 i PMS2) djeluju koordinirano u uzastopnim koracima kako bi započeli popravku DNK nepodudarnosti.[23] In addition, there are Exo1-dependent and Exo1-independent MMR subpathways.[24]
Ostali genski proizvodi uključeni u popravku neusklađenosti (nakon iniciranja MMR gena) kod ljudi uključuju DNK-polimerazu delta, PCNA, RPA, HMGB1, RFC i DNK ligazu I, plus histon i hromatin modificirajući faktori.[25][26]
U određenim okolnostima, MMR put može regrutovati DNK-polimerazu eta, sklonu greškama (POLH). Ovo se dešava u B-limfocitima tokom somatske hipermutacije, gdje se POLH koristi za uvođenje genetičke varijacije u gene antitijela.[27] Međutim, ovaj MMR put sklon greškama može se pokrenuti u drugim tipovima ljudskih ćelija nakon izlaganja genotoksinima [28] i zaista je široko aktivan u različitim ljudskim karcinomima, uzrokujući mutacije koje nose potpis POLH aktivnosti.[29]
MMR i frekvencija mutacija
urediPrepoznavanje i popravljanje neusklađenosti i indela je važno za ćelije jer neuspeh u tome rezultira nestabilčnošću mikrosatelita (MSI) i povećanom spontanom sopom mutacija (mutatorski fenotip). U poređenju sa drugim tipovima karcinoma, rak s nedostatkom MMR-a (MSI) ima vrlo visoku učestalost mutacija, blizu melanoma i raka pluća,[30] tipova raka uzrokovanih velikom izloženošću UV zračenju i mutagenim hemikalijama.
Pored vrlo velikog opterećenja mutacija, nedostaci MMR-a rezultiraju neobičnom distribucijom somatskih mutacija u ljudskom genomu: ovo sugerira da MMR preferencijalno štiti genom bogate euhromatinske regije koje se rano repliciraju.[31] Nasuprot tome, genomski siromašni, kasno replicirajući heteroromatinski genomski regioni pokazuju visoke stope mutacija u mnogim ljudskim tumorima.[32]
Histonska modifikacija H3K36me3, epigenetičkih oznaka aktivnog hromatina, ima sposobnost da regrutuje kompleks MSH2-MSH6 (hMutSα).[33] Konzistentno, regije ljudskog genoma s visokim nivoom H3K36me3 akumuliraju manje mutacija zbog aktivnosti MMR.[29]
Gubitak višestrukih puteva popravka DNK u tumorima
urediNedostatak MMR se često javlja u koordinaciji sa gubitkom drugih gena za popravku DNK.[9] Naprimjer, MMR geni MLH1 i MLH3 također kao što je 11 drugih gena za popravku DNK (kao što je MGMT i mnogi geni puta gena NER) značajno smanjeno u nižem stupnju kao kao i kod astrocitoma višeg stepena, za razliku od normalnog moždanog tkiva.[34] Štaviše, ekspresija MLH1 i MGMT bila je usko povezana u 135 uzoraka raka želuca i činilo se da je gubitak MLH1 i MGMT sinhrono ubrzan tokom progresije tumora.[35]
Nedostatak ekspresije višestrukih gena za popravku DNK često se nalazi kod karcinoma,[9] i može doprinijeti hiljadama mutacija koje se obično nalaze kod karcinoma (pogledajte Frekvencije mutacija kod karcinoma) .
Starenje
urediPopularna ideja, koja nije dobila značajnu eksperimentalnu podršku, je ideja da je mutacija, za razliku od oštećenja DNK, primarni uzrok starenja. Miševi defektni u mutL homologu Pms2 imaju oko 100 puta povećanu učestalost mutacija u svim tkivima, ali izgleda da ne stare brže.[36] Ovi miševi uglavnom pokazuju normalan razvoj i život, osim ranog početka karcinogeneze i muške neplodnosti.
Također pogledajte
urediReference
uredi- ^ Iyer RR, Pluciennik A, Burdett V, Modrich PL (februar 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- ^ Larrea AA, Lujan SA, Kunkel TA (maj 2010). "SnapShot: DNA mismatch repair". Cell. 141 (4): 730–730.e1. doi:10.1016/j.cell.2010.05.002. PMID 20478261. S2CID 26969788.
- ^ Heller RC, Marians KJ (decembar 2006). "Replisome assembly and the direct restart of stalled replication forks". Nature Reviews. Molecular Cell Biology. 7 (12): 932–43. doi:10.1038/nrm2058. PMID 17139333. S2CID 27666329.
- ^ Pluciennik A, Dzantiev L, Iyer RR, Constantin N, Kadyrov FA, Modrich P (septembar 2010). "PCNA function in the activation and strand direction of MutLα endonuclease in mismatch repair". Proceedings of the National Academy of Sciences of the United States of America. 107 (37): 16066–71. doi:10.1073/pnas.1010662107. PMC 2941292. PMID 20713735.
- ^ Kadyrov FA, Dzantiev L, Constantin N, Modrich P (juli 2006). "Endonucleolytic function of MutLalpha in human mismatch repair". Cell. 126 (2): 297–308. doi:10.1016/j.cell.2006.05.039. PMID 16873062. S2CID 15643051.
- ^ Flores-Rozas H, Clark D, Kolodner RD (novembar 2000). "Proliferating cell nuclear antigen and Msh2p-Msh6p interact to form an active mispair recognition complex". Nature Genetics. 26 (3): 375–8. doi:10.1038/81708. PMID 11062484. S2CID 20861705.
- ^ Iyer RR, Pohlhaus TJ, Chen S, Hura GL, Dzantiev L, Beese LS, Modrich P (maj 2008). "The MutSalpha-proliferating cell nuclear antigen interaction in human DNA mismatch repair". The Journal of Biological Chemistry. 283 (19): 13310–9. doi:10.1074/jbc.M800606200. PMC 2423938. PMID 18326858.
- ^ OMIM: 276300
- ^ a b c Bernstein C, Bernstein H (maj 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ a b c Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, et al. (maj 2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, et al. (april 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ Kupčinskaitė-Noreikienė R, Skiecevičienė J, Jonaitis L, Ugenskienė R, Kupčinskas J, Markelis R, et al. (2013). "CpG island methylation of the MLH1, MGMT, DAPK, and CASP8 genes in cancerous and adjacent noncancerous stomach tissues". Medicina. 49 (8): 361–6. doi:10.3390/medicina49080056. PMID 24509146.
- ^ Waki T, Tamura G, Tsuchiya T, Sato K, Nishizuka S, Motoyama T (august 2002). "Promoter methylation status of E-cadherin, hMLH1, and p16 genes in nonneoplastic gastric epithelia". The American Journal of Pathology. 161 (2): 399–403. doi:10.1016/S0002-9440(10)64195-8. PMC 1850716. PMID 12163364.
- ^ Endoh Y, Tamura G, Ajioka Y, Watanabe H, Motoyama T (septembar 2000). "Frequent hypermethylation of the hMLH1 gene promoter in differentiated-type tumors of the stomach with the gastric foveolar phenotype". The American Journal of Pathology. 157 (3): 717–22. doi:10.1016/S0002-9440(10)64584-1. PMC 1949419. PMID 10980110.
- ^ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, et al. (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley" (PDF). Asian Pacific Journal of Cancer Prevention. 13 (8): 4177–81. doi:10.7314/apjcp.2012.13.8.4177. PMID 23098428.
- ^ Chang Z, Zhang W, Chang Z, Song M, Qin Y, Chang F, et al. (januar 2015). "Expression characteristics of FHIT, p53, BRCA2 and MLH1 in families with a history of oesophageal cancer in a region with a high incidence of oesophageal cancer". Oncology Letters. 9 (1): 430–436. doi:10.3892/ol.2014.2682. PMC 4246613. PMID 25436004.
- ^ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". American Journal of Otolaryngology. 32 (6): 528–36. doi:10.1016/j.amjoto.2010.11.005. PMID 21353335.
- ^ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, et al. (oktobar 2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngology–Head and Neck Surgery. 141 (4): 484–90. doi:10.1016/j.otohns.2009.07.007. PMID 19786217. S2CID 8357370.
- ^ Safar AM, Spencer H, Su X, Coffey M, Cooney CA, Ratnasinghe LD, et al. (juni 2005). "Methylation profiling of archived non-small cell lung cancer: a promising prognostic system". Clinical Cancer Research. 11 (12): 4400–5. doi:10.1158/1078-0432.CCR-04-2378. PMID 15958624.
- ^ Rubin H (mart 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ^ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, et al. (februar 2000). "Genetic reconstruction of individual colorectal tumor histories". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (mart 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Pal T, Permuth-Wey J, Sellers TA (august 2008). "A review of the clinical relevance of mismatch-repair deficiency in ovarian cancer". Cancer. 113 (4): 733–42. doi:10.1002/cncr.23601. PMC 2644411. PMID 18543306.
- ^ Goellner EM, Putnam CD, Kolodner RD (august 2015). "Exonuclease 1-dependent and independent mismatch repair". DNA Repair. 32: 24–32. doi:10.1016/j.dnarep.2015.04.010. PMC 4522362. PMID 25956862.
- ^ Li GM (januar 2008). "Mechanisms and functions of DNA mismatch repair". Cell Research. 18 (1): 85–98. doi:10.1038/cr.2007.115. PMID 18157157.
- ^ Li GM (juli 2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair. 19: 48–54. doi:10.1016/j.dnarep.2014.03.027. PMC 4127414. PMID 24767944.
- ^ Chahwan R, Edelmann W, Scharff MD, Roa S (august 2012). "AIDing antibody diversity by error-prone mismatch repair". Seminars in Immunology. 24 (4): 293–300. doi:10.1016/j.smim.2012.05.005. PMC 3422444. PMID 22703640.
- ^ Hsieh P (septembar 2012). "DNA mismatch repair: Dr. Jekyll and Mr. Hyde?". Molecular Cell. 47 (5): 665–6. doi:10.1016/j.molcel.2012.08.020. PMC 3457060. PMID 22980456.
- ^ a b Supek F, Lehner B (juli 2017). "Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes". Cell. 170 (3): 534–547.e23. doi:10.1016/j.cell.2017.07.003. hdl:10230/35343. PMID 28753428.
- ^ Tuna M, Amos CI (novembar 2013). "Genomic sequencing in cancer". Cancer Letters. 340 (2): 161–70. doi:10.1016/j.canlet.2012.11.004. PMC 3622788. PMID 23178448.
- ^ Supek F, Lehner B (maj 2015). "Differential DNA mismatch repair underlies mutation rate variation across the human genome". Nature. 521 (7550): 81–4. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ^ Schuster-Böckler B, Lehner B (august 2012). "Chromatin organization is a major influence on regional mutation rates in human cancer cells". Nature. 488 (7412): 504–7. Bibcode:2012Natur.488..504S. doi:10.1038/nature11273. PMID 22820252. S2CID 205229634.
- ^ Li F, Mao G, Tong D, Huang J, Gu L, Yang W, Li GM (april 2013). "The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα". Cell. 153 (3): 590–600. doi:10.1016/j.cell.2013.03.025. PMC 3641580. PMID 23622243.
- ^ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (decembar 2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neuroscience Letters. 409 (2): 112–7. doi:10.1016/j.neulet.2006.09.038. PMID 17034947. S2CID 54278905.
- ^ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer. 6 (2): 86–95. doi:10.1007/s10120-003-0213-z. PMID 12861399.
- ^ Narayanan L, Fritzell JA, Baker SM, Liskay RM, Glazer PM (april 1997). "Elevated levels of mutation in multiple tissues of mice deficient in the DNA mismatch repair gene Pms2". Proceedings of the National Academy of Sciences of the United States of America. 94 (7): 3122–7. Bibcode:1997PNAS...94.3122N. doi:10.1073/pnas.94.7.3122. PMC 20332. PMID 9096356.
Dopunska literatura
uredi- Hsieh P, Yamane K (2008). "DNA mismatch repair: molecular mechanism, cancer, and ageing". Mechanisms of Ageing and Development. 129 (7–8): 391–407. doi:10.1016/j.mad.2008.02.012. PMC 2574955. PMID 18406444.
- Iyer RR, Pluciennik A, Burdett V, Modrich PL (februar 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews. 106 (2): 302–23. doi:10.1021/cr0404794. PMID 16464007.
- Joseph N, Duppatla V, Rao DN (2006). Prokaryotic DNA mismatch repair. Progress in Nucleic Acid Research and Molecular Biology. 81. str. 1–49. doi:10.1016/S0079-6603(06)81001-9. ISBN 9780125400817. PMID 16891168.
- Yang W (august 2000). "Structure and function of mismatch repair proteins". Mutation Research. 460 (3–4): 245–56. doi:10.1016/s0921-8777(00)00030-6. PMID 10946232.
- Griffiths JF, Gilbert WM, Lewontin RC, Wessler SR, Suzuki DT, Miller JH (2004). An introduction to genetic analysis (8th izd.). New York, NY: Freeman. ISBN 978-0-7167-4939-4.
- Kunkel TA, Erie DA (2005). "DNA mismatch repair". Annual Review of Biochemistry. 74: 681–710. doi:10.1146/annurev.biochem.74.082803.133243. PMID 15952900.
- Friedberg EC, Walker GC, Siede W, Wood RD, Schultz RA, Ellenberger (2005). DNA repair and mutagenesis (2nd izd.). Washington, D.C.: ASM Press. ISBN 978-1-55581-319-2.
Vanjski linkovi
uredi- DNA Repair Arhivirano 12. 2. 2018. na Wayback Machine
- DNA Mismatch Repair na US National Library of Medicine Medical Subject Headings (MeSH)