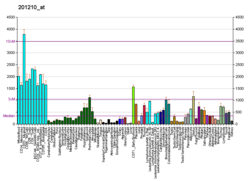
DDX3X
ATP-ovisna RNK-helikaza DDX3X je enzim koji je kod ljudi kodiran genom DDX3X.[5][6][7]
Funkcija
urediProteini DEAD-boksa, karakterizirani konzerviranim motivom Asp-Glu-Ala-Asp (DEAD), navodna su RNK helikaza. Uključeni su u brojne ćelijske procese u izmjeni sekundarne strukture RNK, kao što su eukariotska inicijacija translacije, jedarna i mitohondrijska prerada i ribosomni i splajsosomni sklop. Na osnovu njihovih obrazaca distribucije, vjeruje se da su neki članovi ove porodice uključeni u embriogenezu, spermatogenezu i ćelijski rast i diobu. Ovaj gen kodira protein DEAD kutije, koji specifično komunicira s proteinskim jedrom virusa hepatitisa C, što rezultira promjenom unutarćelijske lokacije. Ovaj gen ima homolog smješten u nerekombinirajućem području hromooma Y. Proteinska sekvenca je 91% identična između ovog gena i Y-vezanog homologa.[7]
Subćelijski promet
urediDDX3X obavlja svoje funkcije u ćelijskom jedru i citoplazmi, izlazeći iz jedra putem eksportina-1 /CRM1 jedarnog eksportnog puta. U početku je objavljeno da je za ovu interakciju neophodan domen helikaze DDX3X, dok su kanonske karakteristike prometnog puta kod ljudi, nisu bili potrebni jedarni ekspotni rni signal (NES) na DDX3X i Ran-GTP koji se vežu za exportin-1,[8] DDX3X od kada je pokazano da vezanje i promet eksportina-1 ne zahtijeva DDX3X domen helikaze i da eksplicitno ovisi o NES i Ran-GTP.[9]
Uloga u kanceru
urediDDX3X je uključen u mnogo različitih tipova karcinoma. Naprimjer, nenormalno se ispoljava u ćelijama epitelnog karcinoma dojke u kojima se njegova ekspresija aktivira pomoću HIF1A tokom hipoksije.[10] Povećana ekspresija DDX3X pomoću HIF1A u hipoksiji pokreće se direktnim vezanjem HIF1A za HIF1A element odgovora, kako je verifikovano imunoprecipitacijom hromatina i testom reportera luciferaze. Budući da na ekspresiju DDX3X utiče aktivnost HIF1A, kolokalizacija ovih proteina je također demonstrirana u MDA-MB-231 ksenotransplantu uzorka tumora.
Izviješteno je da HeLa ćelije DDX3X-a kontroliraju napredovanje ćelijskog ciklusa pomoću ciklina E1.[11] Preciznije, pokazano je da se DDX3X direktno veže za 5´ UTR ciklina E1 i na taj način olakšava translaciju proteina. Pokazano je da povećani nivo proteina cikklin E1 posreduje u tranziciji ulaska u [[S -faza|S-fazu]].
DDX3X utječe na aktivnost preživljavanja, migraciju i proliferaciju melanoma.[12] Ćelije melanoma s niskom ekspresijom DDX3X pokazuju visok migracijski kapacitet, nisku stopu proliferacije i smanjenu osjetljivost na vemurafenib. Iako su ćelije sa visokim ekspresijom DDX3X osjetljive na lijekove, proliferativnije i manje migratorne. Ovi fenotipovi se mogu objasniti translacijskim efektima na faktor transkripcije melanoma MITF.[12]MITF iRNK 5'UTR sadrži složeni RNK regulon (IRES) koji je vezan i aktiviran putem DDX3X. Aktivacija IRES-a dovodi do translacije mITF MITF. Miševi kojima su ubrizgane ćelije melanoma sa deletiranim IRES pokazuju agresivniju progresiju tumora, uključujući povećane metastaze pluća.[12] Zanimljivo je da na DDX3X u melanomu vemurafenib utječe putem neotkrivenog mehanizma transdukcije signala. Nepoznato je kako prisustvo DDX3X regulira prisustvo vemurafeniba. Međutim, smanjeni nivoi DDX3X tokom uzimanja lijekova objašnjavaju razvoj ćelija otpornih na lijekove koji se često otkrivaju s niskom ekspresijom MITF.[12][13][14]
Klinički značaj
urediMutacije gena DDX3X povezane su sa medulloblastomom.[15][16][17] U melanomu je niska ekspresija gena povezana sa lošim preživljavanje bez udaljenih metastaza.[12] Pored toga, nivo iRNK DDX3X je niži u podudarnim biopsijama melanoma nakon recidiva kod pacijenata koji primaju vemurafenib i kod tumora koji napreduju.
Također pogledajte
urediReference
uredi- ^ a b c GRCh38: Ensembl release 89: ENSG00000215301 - Ensembl, maj 2017
- ^ a b c GRCm38: Ensembl release 89: ENSMUSG00000000787 - Ensembl, maj 2017
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Lahn BT, Page DC (oktobar 1997). "Functional coherence of the human Y chromosome". Science. 278 (5338): 675–80. doi:10.1126/science.278.5338.675. PMID 9381176.
- ^ Park SH, Lee SG, Kim Y, Song K (Oct 1998). "Assignment of a human putative RNA helicase gene, DDX3, to human X chromosome bands p11.3→p11.23". Cytogenetics and Cell Genetics. 81 (3–4): 178–9. doi:10.1159/000015022. PMID 9730595.
- ^ a b "Entrez Gene: DDX3X DEAD (Asp-Glu-Ala-Asp) box polypeptide 3, X-linked".
- ^ Yedavalli VS, Neuveut C, Chi YH, Kleiman L, Jeang KT (oktobar 2004). "Requirement of DDX3 DEAD box RNA helicase for HIV-1 Rev-RRE export function". Cell (jezik: engleski). 119 (3): 381–92. doi:10.1016/j.cell.2004.09.029. PMID 15507209.
- ^ Heaton SM, Atkinson SC, Sweeney MN, Yang SN, Jans DA, Borg NA (septembar 2019). "Exportin-1-Dependent Nuclear Export of DEAD-box Helicase DDX3X is Central to its Role in Antiviral Immunity". Cells. 8 (10): 1181. doi:10.3390/cells8101181. PMID 31575075.
- ^ Botlagunta M, Krishnamachary B, Vesuna F, Winnard PT, Bol GM, Patel AH, Raman V (mart 2011). "Expression of DDX3 is directly modulated by hypoxia inducible factor-1 alpha in breast epithelial cells". PLOS ONE. 6 (3): e17563. doi:10.1371/journal.pone.0017563. PMC 3063174. PMID 21448281.
- ^ Lai MC, Chang WC, Shieh SY, Tarn WY (novembar 2010). "DDX3 regulates cell growth through translational control of cyclin E1". Molecular and Cellular Biology. 30 (22): 5444–53. doi:10.1128/MCB.00560-10. PMC 2976371. PMID 20837705.
- ^ a b c d e Phung B, Cieśla M, Sanna A, Guzzi N, Beneventi G, Cao Thi Ngoc P, et al. (juni 2019). "The X-Linked DDX3X RNA Helicase Dictates Translation Reprogramming and Metastasis in Melanoma". Cell Reports. 27 (12): 3573–3586.e7. doi:10.1016/j.celrep.2019.05.069. PMID 31216476.
- ^ Müller J, Krijgsman O, Tsoi J, Robert L, Hugo W, Song C, et al. (decembar 2014). "Low MITF/AXL ratio predicts early resistance to multiple targeted drugs in melanoma". Nature Communications. 5 (1): 5712. doi:10.1038/ncomms6712. PMC 4428333. PMID 25502142.
- ^ Konieczkowski DJ, Johannessen CM, Abudayyeh O, Kim JW, Cooper ZA, Piris A, et al. (juli 2014). "A melanoma cell state distinction influences sensitivity to MAPK pathway inhibitors". Cancer Discovery. 4 (7): 816–27. doi:10.1158/2159-8290.CD-13-0424. PMC 4154497. PMID 24771846.
- ^ Robinson G, Parker M, Kranenburg TA, Lu C, Chen X, Ding L, et al. (august 2012). "Novel mutations target distinct subgroups of medulloblastoma". Nature. 488 (7409): 43–8. doi:10.1038/nature11213. PMC 3412905. PMID 22722829.
- ^ Jones DT, Jäger N, Kool M, Zichner T, Hutter B, Sultan M, et al. (august 2012). "Dissecting the genomic complexity underlying medulloblastoma". Nature. 488 (7409): 100–5. doi:10.1038/nature11284. PMC 3662966. PMID 22832583.
- ^ Pugh TJ, Weeraratne SD, Archer TC, Pomeranz Krummel DA, Auclair D, Bochicchio J, et al. (august 2012). "Medulloblastoma exome sequencing uncovers subtype-specific somatic mutations". Nature. 488 (7409): 106–10. doi:10.1038/nature11329. PMC 3413789. PMID 22820256.
Dopunska literatura
uredi- Li L, Li HS, Pauza CD, Bukrinsky M, Zhao RY (2006). "Roles of HIV-1 auxiliary proteins in viral pathogenesis and host-pathogen interactions". Cell Research. 15 (11–12): 923–34. doi:10.1038/sj.cr.7290370. PMID 16354571.
- Owsianka AM, Patel AH (maj 1999). "Hepatitis C virus core protein interacts with a human DEAD box protein DDX3". Virology. 257 (2): 330–40. doi:10.1006/viro.1999.9659. PMID 10329544.
- Mamiya N, Worman HJ (maj 1999). "Hepatitis C virus core protein binds to a DEAD box RNA helicase". The Journal of Biological Chemistry. 274 (22): 15751–6. doi:10.1074/jbc.274.22.15751. PMID 10336476.
- Yagüe J, Alvarez I, Rognan D, Ramos M, Vázquez J, de Castro JA (juni 2000). "An N-acetylated natural ligand of human histocompatibility leukocyte antigen (HLA)-B39. Classical major histocompatibility complex class I proteins bind peptides with a blocked NH(2) terminus in vivo". The Journal of Experimental Medicine. 191 (12): 2083–92. doi:10.1084/jem.191.12.2083. PMC 2193201. PMID 10859333.
- Kim YS, Lee SG, Park SH, Song K (oktobar 2001). "Gene structure of the human DDX3 and chromosome mapping of its related sequences". Molecules and Cells. 12 (2): 209–14. PMID 11710523.
- Li J, Hawkins IC, Harvey CD, Jennings JL, Link AJ, Patton JG (novembar 2003). "Regulation of alternative splicing by SRrp86 and its interacting proteins". Molecular and Cellular Biology. 23 (21): 7437–47. doi:10.1128/MCB.23.21.7437-7447.2003. PMC 207616. PMID 14559993.
- Shu H, Chen S, Bi Q, Mumby M, Brekken DL (mart 2004). "Identification of phosphoproteins and their phosphorylation sites in the WEHI-231 B lymphoma cell line". Molecular & Cellular Proteomics. 3 (3): 279–86. doi:10.1074/mcp.D300003-MCP200. PMID 14729942.
- Bouwmeester T, Bauch A, Ruffner H, Angrand PO, Bergamini G, Croughton K, Cruciat C, Eberhard D, Gagneur J, Ghidelli S, Hopf C, Huhse B, Mangano R, Michon AM, Schirle M, Schlegl J, Schwab M, Stein MA, Bauer A, Casari G, Drewes G, Gavin AC, Jackson DB, Joberty G, Neubauer G, Rick J, Kuster B, Superti-Furga G (februar 2004). "A physical and functional map of the human TNF-alpha/NF-kappa B signal transduction pathway". Nature Cell Biology. 6 (2): 97–105. doi:10.1038/ncb1086. PMID 14743216.
- Yedavalli VS, Neuveut C, Chi YH, Kleiman L, Jeang KT (oktobar 2004). "Requirement of DDX3 DEAD box RNA helicase for HIV-1 Rev-RRE export function". Cell. 119 (3): 381–92. doi:10.1016/j.cell.2004.09.029. PMID 15507209.
- Dayton AI (oktobar 2004). "Within you, without you: HIV-1 Rev and RNA export". Retrovirology. 1: 35. doi:10.1186/1742-4690-1-35. PMC 526764. PMID 15516266.
- Krishnan V, Zeichner SL (decembar 2004). "Alterations in the expression of DEAD-box and other RNA binding proteins during HIV-1 replication". Retrovirology. 1: 42. doi:10.1186/1742-4690-1-42. PMC 543576. PMID 15588285.
- Rush J, Moritz A, Lee KA, Guo A, Goss VL, Spek EJ, Zhang H, Zha XM, Polakiewicz RD, Comb MJ (januar 2005). "Immunoaffinity profiling of tyrosine phosphorylation in cancer cells". Nature Biotechnology. 23 (1): 94–101. doi:10.1038/nbt1046. PMID 15592455.
- Tao WA, Wollscheid B, O'Brien R, Eng JK, Li XJ, Bodenmiller B, Watts JD, Hood L, Aebersold R (august 2005). "Quantitative phosphoproteome analysis using a dendrimer conjugation chemistry and tandem mass spectrometry". Nature Methods. 2 (8): 591–8. doi:10.1038/nmeth776. PMID 16094384.
- Gevaert K, Staes A, Van Damme J, De Groot S, Hugelier K, Demol H, Martens L, Goethals M, Vandekerckhove J (septembar 2005). "Global phosphoproteome analysis on human HepG2 hepatocytes using reversed-phase diagonal LC". Proteomics. 5 (14): 3589–99. doi:10.1002/pmic.200401217. PMID 16097034.
- Chang PC, Chi CW, Chau GY, Li FY, Tsai YH, Wu JC, Wu Lee YH (mart 2006). "DDX3, a DEAD box RNA helicase, is deregulated in hepatitis virus-associated hepatocellular carcinoma and is involved in cell growth control". Oncogene. 25 (14): 1991–2003. doi:10.1038/sj.onc.1209239. PMID 16301996.